library(tidyverse)#> ── Attaching core tidyverse packages ───────────────────── tidyverse 2.0.0 ──#> ✔ dplyr 1.1.0.9000 ✔ readr 2.1.4#> ✔ forcats 1.0.0 ✔ stringr 1.5.0#> ✔ ggplot2 3.4.1 ✔ tibble 3.1.8#> ✔ lubridate 1.9.2 ✔ tidyr 1.3.0#> ✔ purrr 1.0.1#> ── Conflicts ─────────────────────────────────────── tidyverse_conflicts() ──#> ✖ dplyr::filter() masks stats::filter()#> ✖ dplyr::lag() masks stats::lag()#> ℹ Use the conflicted package (<http://conflicted.r-lib.org/>) to force all#> conflicts to become errors
Capítulo 1. Visualización de datos
Este trabajo se ha traducido utilizando IA. Agradecemos tus opiniones y comentarios: translation-feedback@oreilly.com
Introducción
"El simple gráfico ha aportado más información a la mente del analista de datos que cualquier otro dispositivo". -John Tukey
R tiene varios sistemas para hacer gráficos, pero ggplot2 es uno de los más elegantes y versátiles. ggplot2 implementa la gramática de los gráficos, un sistema coherente para describir y construir gráficos. Con ggplot2, puedes hacer más cosas más rápido aprendiendo un sistema y aplicándolo en muchos sitios.
Este capítulo te enseñará a visualizar tus datos utilizando ggplot2. Empezaremos creando un simple gráfico de dispersión y lo utilizaremos para introducir los mapeados estéticos y los objetos geométricos, los bloques de construcción fundamentales de ggplot2. A continuación, te guiaremos a través de la visualización de distribuciones de variables individuales, así como de la visualización de relaciones entre dos o más variables. Terminaremos guardando tus gráficos y con consejos para solucionar problemas.
Requisitos previos
Este capítulo se centra en ggplot2, uno de los paquetes centrales del tidyverse. Para acceder a los conjuntos de datos, páginas de ayuda y funciones utilizadas en este capítulo, carga el tidyverse ejecutando:
Esa única línea de código carga el núcleo del tidyverse, los paquetes que utilizarás en casi todos los análisis de datos. También te indica qué funciones del tidyverse entran en conflicto con funciones de R base (o de otros paquetes que hayas cargado).1
Si ejecutas este código y obtienes el mensaje de error there is no package called 'tidyverse', primero tendrás que instalarlo y luego ejecutar library() una vez más:
install.packages("tidyverse")library(tidyverse)
Sólo tienes que instalar un paquete una vez, pero tienes que cargarlo cada vez que inicies una nueva sesión.
Además de tidyverse, utilizaremos el paquete palmerpenguins, que incluye el conjunto de datos penguins con medidas corporales de pingüinos de tres islas del archipiélago Palmer, y el paquete ggthemes, que ofrece una paleta de colores segura para daltónicos.
library(palmerpenguins)library(ggthemes)
Primeros pasos
¿Los pingüinos con aletas más largas pesan más o menos que los pingüinos con aletas más cortas? Probablemente ya tengas una respuesta, pero intenta que sea precisa. ¿Cómo es la relación entre la longitud de las aletas y la masa corporal? ¿Es positiva? ¿Negativa? ¿lineal? ¿No lineal? ¿Varía la relación según la especie del pingüino? ¿Y según la isla en la que vive el pingüino? Creemos visualizaciones que podamos utilizar para responder a estas preguntas.
Los pingüinos Marco de datos
Puedes comprobar tus respuestas a estas preguntas con el marco de datos penguins que se encuentra en palmerpenguins (alias palmerpenguins::penguins). Un marco de datos es una colección rectangular de variables (en las columnas) y observaciones (en las filas). penguins contiene 344 observaciones recogidas y puestas a disposición por la Dra. Kristen Gorman y el LTER de la Estación Palmer, Antártida.2
Para facilitar la discusión, definamos algunos términos:
- Variable
- Valor
El estado de una variable cuando la mides. El valor de una variable puede cambiar de una medición a otra.
- Observación
Conjunto de mediciones realizadas en condiciones similares (normalmente realizas todas las mediciones de una observación al mismo tiempo y sobre el mismo objeto). Una observación contendrá varios valores, cada uno asociado a una variable diferente. A veces nos referiremos a una observación como un punto de datos.
- Datos tabulares
Un conjunto de valores, cada uno asociado a una variable y a una observación. Los datos tabulares están ordenados si cada valor se coloca en su propia "celda", cada variable en su propia columna y cada observación en su propia fila.
En este contexto, una variable se refiere a un atributo de todos los pingüinos, y una observación se refiere a todos los atributos de un solo pingüino.
Escribe el nombre del marco de datos en la consola, y R imprimirá una vista previa de su contenido. Observa que encima de esta vista previa pone tibble. En el tidyverse, utilizamos marcos de datos especiales llamados tibbles, sobre los que aprenderás pronto.
penguins#> # A tibble: 344 × 8#> species island bill_length_mm bill_depth_mm flipper_length_mm#> <fct> <fct> <dbl> <dbl> <int>#> 1 Adelie Torgersen 39.1 18.7 181#> 2 Adelie Torgersen 39.5 17.4 186#> 3 Adelie Torgersen 40.3 18 195#> 4 Adelie Torgersen NA NA NA#> 5 Adelie Torgersen 36.7 19.3 193#> 6 Adelie Torgersen 39.3 20.6 190#> # … with 338 more rows, and 3 more variables: body_mass_g <int>, sex <fct>,#> # year <int>
Este marco de datos contiene ocho columnas. Para una vista alternativa, en la que puedas ver todas las variables y las primeras observaciones de cada variable, utiliza glimpse(). O, si estás en RStudio, ejecuta View(penguins) para abrir un visor de datos interactivo.
glimpse(penguins)#> Rows: 344#> Columns: 8#> $ species <fct> Adelie, Adelie, Adelie, Adelie, Adelie, Adelie, A…#> $ island <fct> Torgersen, Torgersen, Torgersen, Torgersen, Torge…#> $ bill_length_mm <dbl> 39.1, 39.5, 40.3, NA, 36.7, 39.3, 38.9, 39.2, 34.…#> $ bill_depth_mm <dbl> 18.7, 17.4, 18.0, NA, 19.3, 20.6, 17.8, 19.6, 18.…#> $ flipper_length_mm <int> 181, 186, 195, NA, 193, 190, 181, 195, 193, 190, …#> $ body_mass_g <int> 3750, 3800, 3250, NA, 3450, 3650, 3625, 4675, 347…#> $ sex <fct> male, female, female, NA, female, male, female, m…#> $ year <int> 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2007, 2…
Entre las variables de penguins están:
speciesLa especie de un pingüino (Adelia, Barbijo o Papúa)
flipper_length_mmLa longitud de la aleta de un pingüino, en milímetros
body_mass_gLa masa corporal de un pingüino, en gramos
Para saber más sobre penguins, abre su página de ayuda ejecutando ?penguins.
Objetivo final
Nuestro objetivo final en este capítulo es recrear la siguiente visualización que muestra la relación entre las longitudes de las aletas y las masas corporales de estos pingüinos, teniendo en cuenta la especie del pingüino.
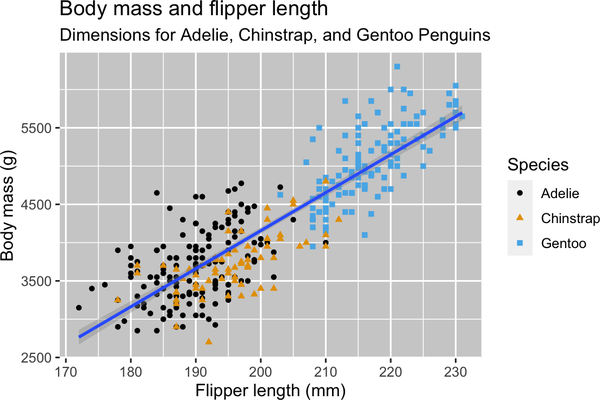
Crear un ggplot
Vamos a recrear esta trama paso a paso.
Con ggplot2, comienzas un gráfico con la función ggplot()definiendo un objeto de gráfico al que luego añades capas. El primer argumento de ggplot() es el conjunto de datos que se utilizará en el gráfico, así que ggplot(data = penguins) crea un gráfico vacío que está preparado para mostrar los datos de penguins, pero como aún no le hemos dicho cómo visualizarlos, por ahora está vacío. No es un gráfico muy emocionante, pero puedes considerarlo como un lienzo vacío en el que pintarás las capas restantes de tu gráfico.
ggplot(data=penguins)

A continuación, tenemos que decir ggplot() cómo se representará visualmente la información de nuestros datos. El argumento mapping de la función ggplot() define cómo se asignan las variables de tu conjunto de datos a las propiedades visuales(estética) de tu gráfico. El argumento mapping siempre se define en la función aes() y los argumentos x y y de la función aes() especifican qué variables asignar a los ejes X e Y. Por ahora, sólo mapearemos la longitud de la aleta a la estética x y la masa corporal a la estética y. ggplot2 busca las variables mapeadas en el argumento data, en este caso, penguins.
El siguiente gráfico muestra el resultado de añadir estas correspondencias.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))
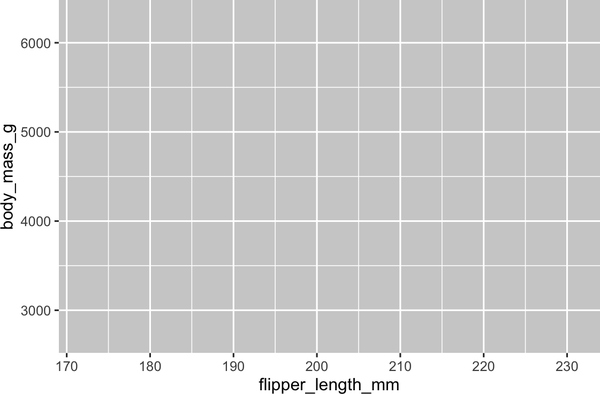
Nuestro lienzo vacío tiene ahora más estructura: está claro dónde se mostrarán las longitudes de las aletas (en el eje x) y dónde se mostrarán las masas corporales (en el eje y). Pero los propios pingüinos aún no aparecen en el gráfico. Esto se debe a que aún no hemos articulado, en nuestro código, cómo representar las observaciones de nuestro marco de datos en nuestro gráfico.
Para ello, necesitamos definir un geom: el objeto geométrico que utiliza un gráfico para representar los datos. Estos objetos geométricos están disponibles en ggplot2 con funciones que empiezan por geom_. A menudo se describen los gráficos por el tipo de geometría que utilizan. Por ejemplo, los gráficos de barras utilizan geomorfos de barras (geom_bar()), los gráficos de líneas utilizan geoms de líneas (geom_line()), los boxplots utilizan geoms de boxplot (geom_boxplot()), los gráficos de dispersión utilizan puntos (geom_point()), etc.
La función geom_point() añade una capa de puntos a tu gráfico, lo que crea un gráfico de dispersión. ggplot2 viene con muchas funciones geom, y cada una añade un tipo diferente de capa a un gráfico. Aprenderás un montón de geoms a lo largo del libro, sobre todo en el Capítulo 9.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_point()#> Warning: Removed 2 rows containing missing values (`geom_point()`).
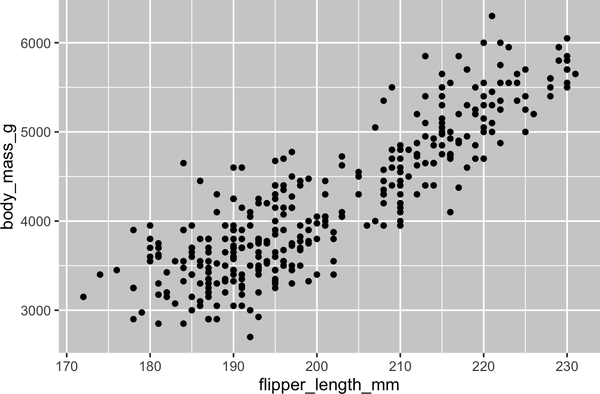
Ahora tenemos algo que se parece a lo que podríamos considerar un "diagrama de dispersión". Todavía no coincide con nuestro gráfico del "objetivo final", pero utilizando este gráfico podemos empezar a responder a la pregunta que motivó nuestra exploración: "¿Cómo es la relación entre la longitud de las aletas y la masa corporal?". La relación parece ser positiva (a medida que aumenta la longitud de las aletas, también lo hace la masa corporal), bastante lineal (los puntos se agrupan en torno a una línea en lugar de una curva) y moderadamente fuerte (no hay demasiada dispersión en torno a dicha línea). Los pingüinos con aletas más largas suelen ser más grandes en términos de masa corporal.
Antes de añadir más capas a esta trama, detengámonos un momento y repasemos el mensaje de advertencia que recibimos:
Eliminadas 2 filas que contenían valores perdidos (
geom_point()).
Aparece este mensaje porque hay dos pingüinos en nuestro conjunto de datos a los que les faltan los valores de masa corporal y/o longitud de las aletas, y ggplot2 no tiene forma de representarlos en el gráfico sin ambos valores. Al igual que R, ggplot2 se adhiere a la filosofía de que los valores que faltan nunca deben desaparecer silenciosamente. Este tipo de advertencia es probablemente uno de los más comunes que verás cuando trabajes con datos reales: los valores perdidos son un problema habitual, y aprenderás más sobre ellos a lo largo del libro, sobre todo en el capítulo 18. En los demás gráficos de este capítulo suprimiremos esta advertencia para que no se imprima junto a cada uno de los gráficos que hagamos.
Añadir estética y capas
Los gráficos de dispersión son útiles para mostrar la relación entre dos variables numéricas, pero siempre es buena idea ser escéptico ante cualquier relación aparente entre dos variables y preguntarse si puede haber otras variables que expliquen o cambien la naturaleza de esta relación aparente. Por ejemplo, ¿la relación entre la longitud de las aletas y la masa corporal difiere según la especie? Incorporemos especies a nuestro gráfico y veamos si esto revela alguna idea adicional sobre la relación aparente entre estas variables. Lo haremos representando las especies con puntos de distinto color.
Para conseguirlo, ¿habrá que modificar la estética o la geom? Si has adivinado "en el mapeo estético, dentro de aes()", ¡ya le estás cogiendo el truco a crear visualizaciones de datos con ggplot2! Y si no, no te preocupes. A lo largo del libro harás muchos más ggplots y tendrás muchas más oportunidades de comprobar tu intuición mientras los haces.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g,color=species))+geom_point()
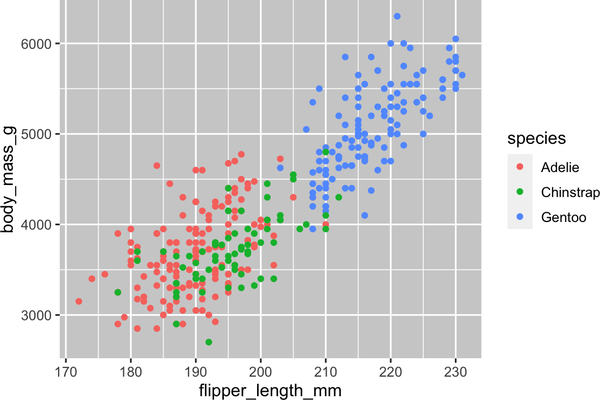
Cuando se asigna una variable categórica a una estética, ggplot2 asignará automáticamente un valor único de la estética (aquí un color único) a cada nivel único de la variable (cada una de las tres especies), proceso conocido como escalado. ggplot2 también añadirá una leyenda que explique qué valores corresponden a qué niveles.
Ahora vamos a añadir una capa más: una curva suave que muestre la relación entre la masa corporal y la longitud de las aletas. Antes de continuar, consulta el código anterior y piensa cómo podemos añadir esto a nuestro gráfico existente.
Como se trata de un nuevo objeto geométrico que representa nuestros datos, añadiremos un nuevo geom como capa sobre nuestro geom de puntos: geom_smooth(). Y especificaremos que queremos dibujar la línea de mejor ajuste basándonos en un linear model con method = "lm".
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g,color=species))+geom_point()+geom_smooth(method="lm")
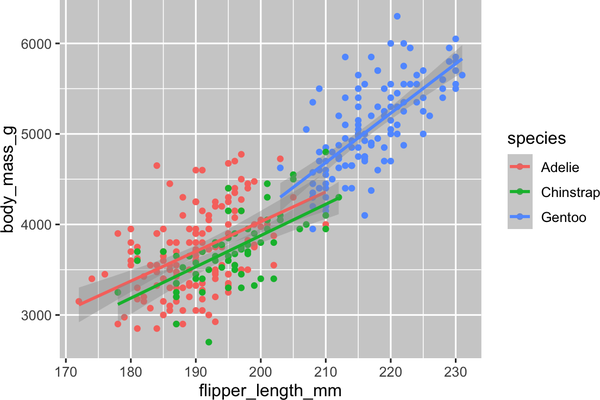
Hemos añadido líneas con éxito, pero este gráfico no se parece al gráfico de "Objetivo final", que sólo tiene una línea para todo el conjunto de datos, en lugar de líneas separadas para cada una de las especies de pingüinos.
Cuando se definen mapeados estéticos en ggplot()a nivel global, se transmiten a cada una de las capas geom subsiguientes del gráfico. Sin embargo, cada función geom en ggplot2 también puede tomar un argumento mapping, que permite mapeados estéticos a nivel local que se añaden a los heredados del nivel global. Como queremos que los puntos se coloreen en función de la especie, pero no queremos que las líneas se separen para ellos, debemos especificar color = species para geom_point() únicamente.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_point(mapping=aes(color=species))+geom_smooth(method="lm")
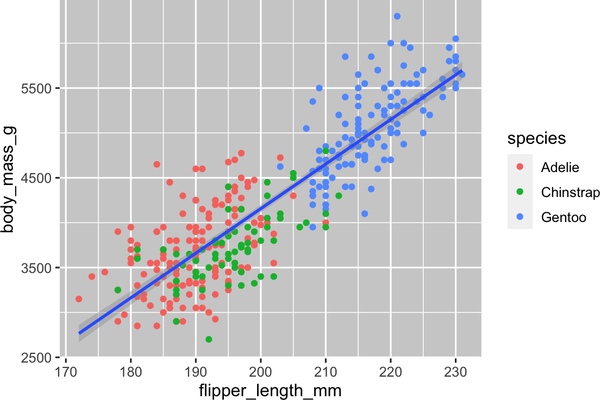
¡Voilà! Tenemos algo que se parece mucho a nuestro objetivo final, aunque todavía no es perfecto. Aún tenemos que utilizar formas diferentes para cada especie de pingüinos y mejorar las etiquetas.
En general, no es buena idea representar la información utilizando sólo colores en un gráfico, ya que las personas perciben los colores de forma diferente debido al daltonismo u otras diferencias de visión cromática. Por lo tanto, además del color, podemos mapear species a la estética shape.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_point(mapping=aes(color=species,shape=species))+geom_smooth(method="lm")
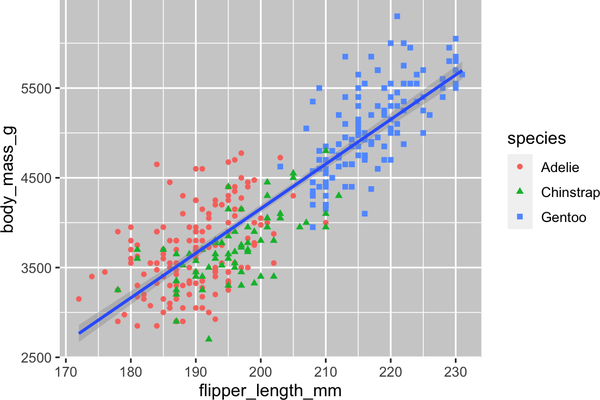
Observa que la leyenda también se actualiza automáticamente para reflejar las diferentes formas de los puntos.
Por último, podemos mejorar las etiquetas de nuestro gráfico utilizando la función labs() en una nueva capa. Algunos de los argumentos de labs() pueden ser autoexplicativos: title añade un título, y subtitle añade un subtítulo al gráfico. Otros argumentos coinciden con los mapeados estéticos: x es la etiqueta del eje x, y es la etiqueta del eje y, y color y shape definen la etiqueta de la leyenda. Además, podemos mejorar la paleta de colores para que sea segura para daltónicos con la función scale_color_colorblind() del paquete ggthemes.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_point(aes(color=species,shape=species))+geom_smooth(method="lm")+labs(title="Body mass and flipper length",subtitle="Dimensions for Adelie, Chinstrap, and Gentoo Penguins",x="Flipper length (mm)",y="Body mass (g)",color="Species",shape="Species")+scale_color_colorblind()
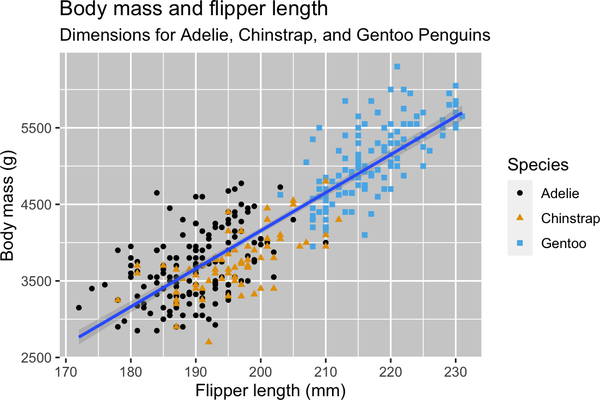
¡Por fin tenemos una parcela que se ajusta perfectamente a nuestro "objetivo final"!
Ejercicios
¿Cuántas filas hay en
penguins? ¿Cuántas columnas?¿Qué describe la variable
bill_depth_mmdel marco de datospenguins? Lee la ayuda de?penguinspara averiguarlo.Haz un gráfico de dispersión de
bill_depth_mmfrente abill_length_mm. Es decir, haz un diagrama de dispersión conbill_depth_mmen el eje y ybill_length_mmen el eje x. Describe la relación entre estas dos variables.¿Qué ocurre si haces un gráfico de dispersión de
speciesfrente abill_depth_mm? ¿Cuál sería una mejor elección de geom?-
¿Por qué da error lo siguiente y cómo lo solucionarías?
ggplot(data=penguins)+geom_point() ¿Qué hace el argumento
na.rmengeom_point()? ¿Cuál es el valor por defecto del argumento? Crea un gráfico de dispersión en el que utilices correctamente este argumento establecido enTRUE.Añade la siguiente leyenda al gráfico que hiciste en el ejercicio anterior: "Los datos proceden del paquete palmerpenguins". Pista: Echa un vistazo a la documentación de
labs().-
Vuelve a crear la siguiente visualización. ¿A qué estética debe asignarse
bill_depth_mm? ¿Y debe mapearse a nivel global o a nivel geom?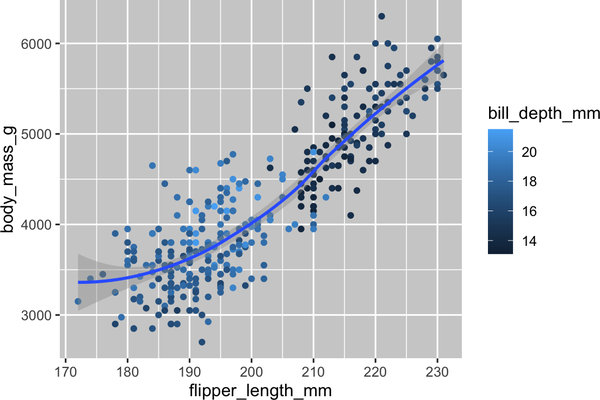
-
Ejecuta este código mentalmente y predice cómo será el resultado. Después, ejecuta el código en R y comprueba tus predicciones.
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g,color=island))+geom_point()+geom_smooth(se=FALSE) -
¿Tendrán estos dos gráficos un aspecto diferente? ¿Por qué?
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_point()+geom_smooth()ggplot()+geom_point(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_smooth(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))
Llamadas ggplot2
A medida que avancemos desde estas secciones introductorias, pasaremos a una expresión más concisa del código ggplot2. Hasta ahora hemos sido muy explícitos, lo que resulta útil cuando estás aprendiendo:
ggplot(data=penguins,mapping=aes(x=flipper_length_mm,y=body_mass_g))+geom_point()
Normalmente, el primero o los dos primeros argumentos de una función son tan importantes que deberías sabértelos de memoria. Los dos primeros argumentos de ggplot() son data y mapping; en el resto del libro, no proporcionaremos esos nombres. Eso ahorra teclear y, al reducir la cantidad de texto extra, hace que sea más fácil ver lo que es diferente entre parcelas. Se trata de una cuestión de programación realmente importante, a la que volveremos en el Capítulo 25.
Reescribiendo la trama anterior de forma más concisa se obtiene:
ggplot(penguins,aes(x=flipper_length_mm,y=body_mass_g))+geom_point()
En el futuro, también conocerás la tubería, |>, que te permitirá crear esa trama con:
penguins|>ggplot(aes(x=flipper_length_mm,y=body_mass_g))+geom_point()
Visualizar distribuciones
La forma de visualizar la distribución de una variable depende del tipo de variable: categórica o numérica.
Una variable categórica
Una variable es categórica si sólo puede tomar uno de un pequeño conjunto de valores. Para examinar la distribución de una variable categórica, puedes utilizar un gráfico de barras. La altura de las barras muestra cuántas observaciones se produjeron con cada valor x.
ggplot(penguins,aes(x=species))+geom_bar()
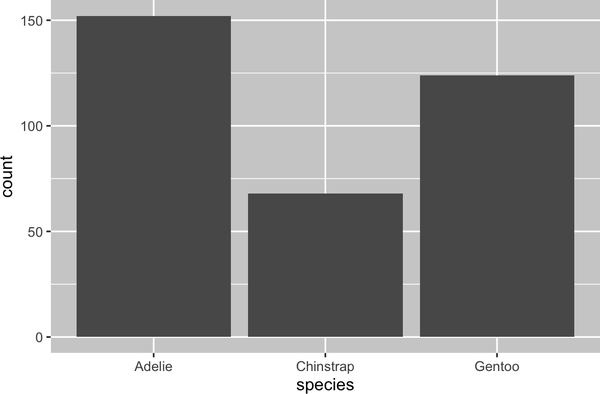
En los gráficos de barras de variables categóricas con niveles no ordenados, como el pingüino anterior species, a menudo es preferible reordenar las barras en función de sus frecuencias. Para ello es necesario transformar la variable en un factor (la forma en que R trata los datos categóricos) y luego reordenar los niveles de ese factor.
ggplot(penguins,aes(x=fct_infreq(species)))+geom_bar()
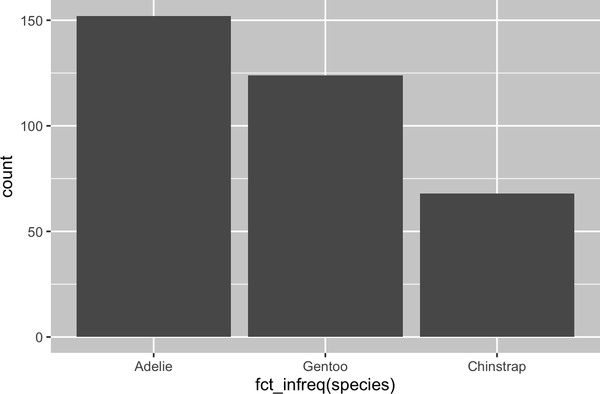
Aprenderás más sobre factores y funciones para tratar con factores (como fct_infreq()) en el capítulo 16.
Una variable numérica
Una variable es numérica (o cuantitativa) si puede adoptar una amplia gama de valores numéricos y es sensato sumar, restar o sacar medias con esos valores. Las variables numéricas pueden ser continuas o discretas.
Una visualización muy utilizada para las distribuciones de variables continuas es el histograma.
ggplot(penguins,aes(x=body_mass_g))+geom_histogram(binwidth=200)
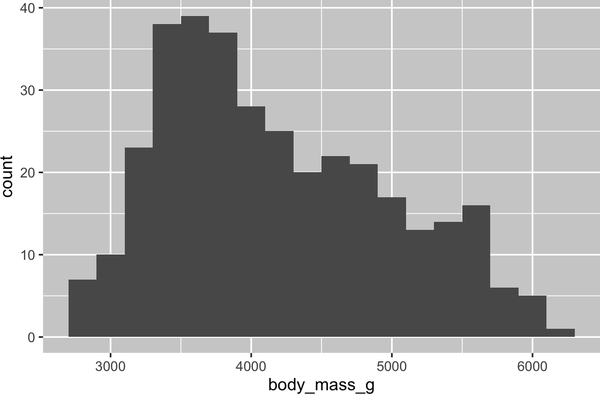
Un histograma divide el eje x en intervalos equidistantes y, a continuación, utiliza la altura de una barra para mostrar el número de observaciones que caen en cada intervalo. En el gráfico anterior, la barra más alta muestra que 39 observaciones tienen un valor body_mass_g entre 3.500 y 3.700 gramos, que son los perímetros izquierdo y derecho de la barra.
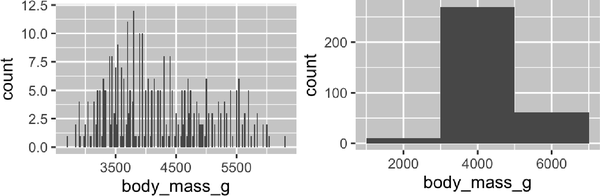
Puedes establecer la anchura de los intervalos en un histograma con el argumento binwidth, que se mide en las unidades de la variable x. Siempre debes explorar una variedad de valores de binwidth cuando trabajes con histogramas, ya que diferentes valores de binwidth pueden revelar patrones diferentes. En los siguientes gráficos, un binwidth de 20 es demasiado estrecho, lo que da lugar a demasiadas barras, dificultando la determinación de la forma de la distribución. Del mismo modo, un binwidth de 2.000 es demasiado alto, lo que hace que todos los datos se dividan en sólo tres barras y también dificulta la determinación de la forma de la distribución. Un binwidth de 200 proporciona un equilibrio razonable.
ggplot(penguins,aes(x=body_mass_g))+geom_histogram(binwidth=20)ggplot(penguins,aes(x=body_mass_g))+geom_histogram(binwidth=2000)
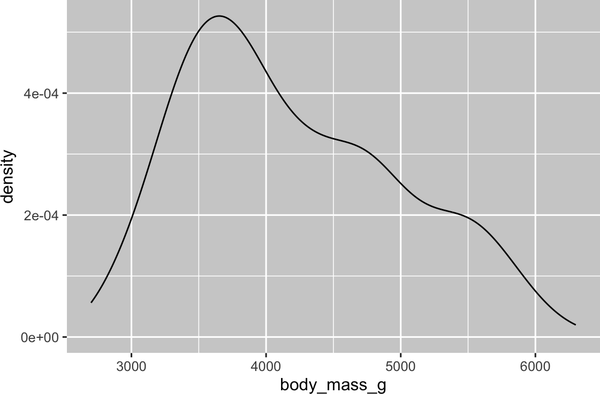
Una visualización alternativa para las distribuciones de variables numéricas es un gráfico de densidad. Un gráfico de densidad es una versión suavizada de un histograma y una alternativa práctica, sobre todo para datos continuos que proceden de una distribución suave subyacente. No vamos a entrar en cómo geom_density() estima la densidad (puedes leer más sobre ello en la documentación de la función), pero vamos a explicar cómo se dibuja la curva de densidad con una analogía. Imagina un histograma hecho con bloques de madera. Luego, imagina que dejas caer sobre él una cuerda de espaguetis cocidos. La forma que adoptarán los espaguetis colocados sobre los bloques puede considerarse la forma de la curva de densidad. Muestra menos detalles que un histograma, pero puede facilitar la comprensión rápida de la forma de la distribución, sobre todo en lo que respecta a las modas y la asimetría.
ggplot(penguins,aes(x=body_mass_g))+geom_density()#> Warning: Removed 2 rows containing non-finite values (`stat_density()`).
Ejercicios
Haz un gráfico de barras de
speciesdepenguins, en el que asignesspeciesa la estéticay. ¿En qué se diferencia este gráfico?-
¿En qué se diferencian los dos gráficos siguientes? ¿Qué estética,
colorofill, es más útil para cambiar el color de las barras?ggplot(penguins,aes(x=species))+geom_bar(color="red")ggplot(penguins,aes(x=species))+geom_bar(fill="red") ¿Qué hace el argumento
binsengeom_histogram()¿para qué sirve?Haz un histograma de la variable
caraten el conjunto de datosdiamondsque está disponible cuando cargas el paquete tidyverse. Experimenta con diferentes valores debinwidth. ¿Qué valor revela los patrones más interesantes?
Visualizar las relaciones
Para visualizar una relación necesitamos tener al menos dos variables mapeadas en la estética de un gráfico. En las siguientes secciones conocerás los trazados más utilizados para visualizar relaciones entre dos o más variables y los geoms que se utilizan para crearlos.
Una variable numérica y otra categórica
Para visualizar la relación entre una variable numérica y una categórica, podemos utilizar gráficos de caja uno al lado del otro. Un diagrama de cajas es un tipo de abreviatura visual de las medidas de posición (percentiles) que describen una distribución. También es útil para identificar posibles valores atípicos. Como se muestra en la Figura 1-1, cada diagrama de caja consta de:
Un recuadro que indica el rango de la mitad media de los datos, una distancia conocida como rango intercuartílico (IQR), que va desde el percentil 25 de la distribución hasta el percentil 75. En el centro del recuadro hay una línea que muestra la mediana, es decir, el percentil 50, de la distribución. Estas tres líneas te dan una idea de la dispersión de la distribución y de si ésta es simétrica respecto a la mediana o está sesgada hacia un lado.
Puntos visuales que muestran observaciones que caen a más de 1,5 veces el IQR de cualquiera de los perímetros de la caja. Estos puntos periféricos son inusuales, por lo que se representan individualmente.
Una línea (o bigote) que se extiende desde cada extremo de la caja y llega hasta el punto más alejado no atípico de la distribución.
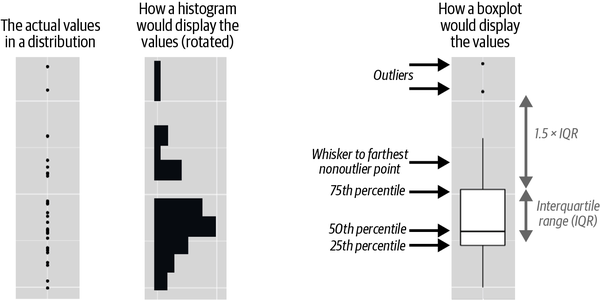
Figura 1-1. Diagrama que muestra cómo se crea un diagrama de caja.
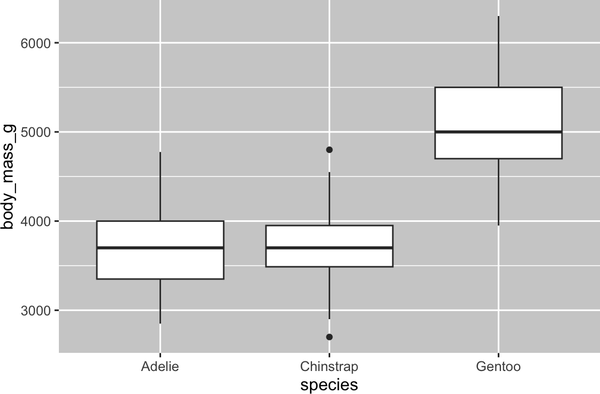
Echemos un vistazo a la distribución de la masa corporal por especies utilizando geom_boxplot():
ggplot(penguins,aes(x=species,y=body_mass_g))+geom_boxplot()
Alternativamente, podemos hacer gráficos de densidad con geom_density():
ggplot(penguins,aes(x=body_mass_g,color=species))+geom_density(linewidth=0.75)
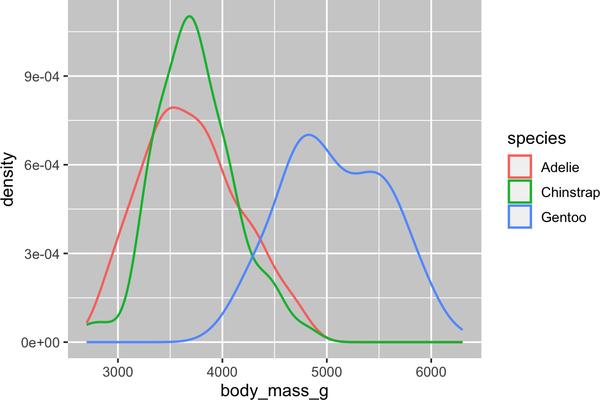
También hemos personalizado el grosor de las líneas utilizando el argumento linewidth para que destaquen un poco más sobre el fondo.
Además, podemos asignar species a las estéticas color y fill y utilizar la estética alpha para añadir transparencia a las curvas de densidad rellenas. Esta estética toma valores entre 0 (completamente transparente) y 1 (completamente opaco). En el siguiente gráfico está ajustada a 0,5:
ggplot(penguins,aes(x=body_mass_g,color=species,fill=species))+geom_density(alpha=0.5)
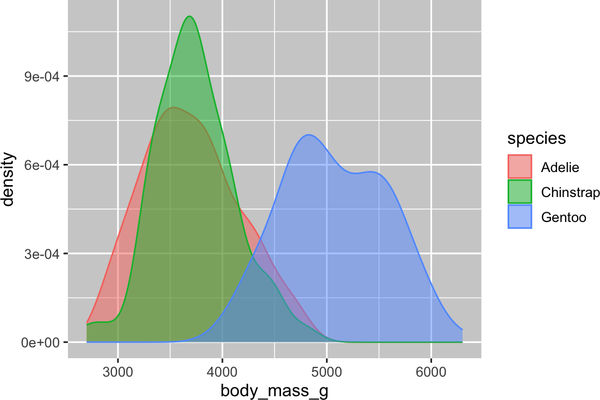
Fíjate en la terminología que hemos utilizado aquí:
- Mapeamos variables a estéticas si queremos que el atributo visual representado por esa estética varíe en función de los valores de esa variable.
- Si no, fijamos el valor de una estética.
Dos variables categóricas
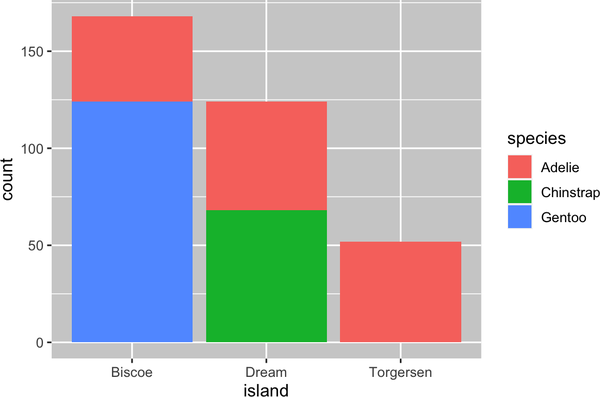
Podemos utilizar gráficos de barras apiladas para visualizar la relación entre dos variables categóricas. Por ejemplo, los dos gráficos de barras apiladas siguientes muestran la relación entre island y species, o, en concreto, visualizan la distribución de species dentro de cada isla.
El primer gráfico muestra las frecuencias de cada especie de pingüinos en cada isla. El gráfico de frecuencias muestra que hay el mismo número de Adelia en cada isla, pero no tenemos una buena idea del equilibrio porcentual dentro de cada isla.
ggplot(penguins,aes(x=island,fill=species))+geom_bar()
El segundo gráfico es un gráfico de frecuencias relativas, creado configurando position = "fill" en el geom, y es más útil para comparar distribuciones de especies entre islas, ya que no se ve afectado por el número desigual de pingüinos en las islas. Con este gráfico podemos ver que todos los pingüinos papúa viven en la isla de Biscoe y constituyen aproximadamente el 75% de los pingüinos de esa isla, todos los barbijos viven en la isla del Sueño y constituyen aproximadamente el 50% de los pingüinos de esa isla, y los Adelia viven en las tres islas y constituyen todos los pingüinos de Torgersen.
ggplot(penguins,aes(x=island,fill=species))+geom_bar(position="fill")
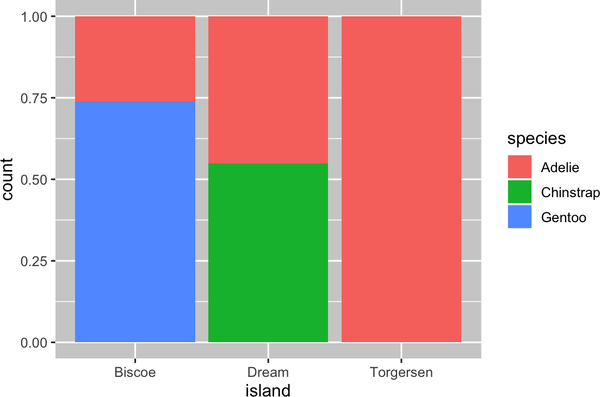
Al crear estos gráficos de barras, asignamos la variable que se separará en barras a la estética x, y la variable que cambiará los colores dentro de las barras a la estética fill.
Dos variables numéricas
Hasta ahora has aprendido sobre gráficos de dispersión (creados con geom_point()) y las curvas suaves (creadas con geom_smooth()) para visualizar la relación entre dos variables numéricas. Un gráfico de dispersión es probablemente el gráfico más utilizado para visualizar la relación entre dos variables numéricas.
ggplot(penguins,aes(x=flipper_length_mm,y=body_mass_g))+geom_point()
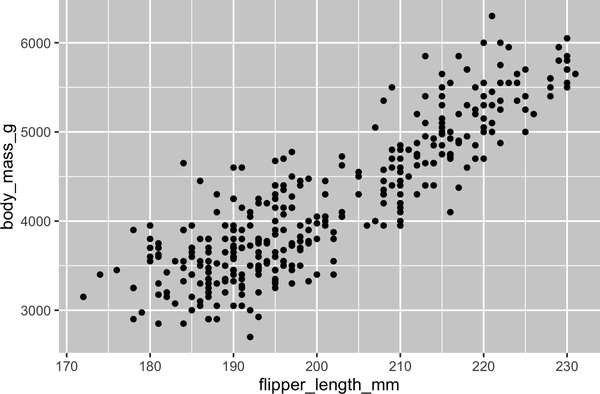
Tres o más variables
Como vimos en "Añadir estéticas y capas", podemos incorporar más variables a un gráfico asignándolas a estéticas adicionales. Por ejemplo, en el siguiente gráfico de dispersión los colores de los puntos representan especies, y las formas de los puntos representan islas:
ggplot(penguins,aes(x=flipper_length_mm,y=body_mass_g))+geom_point(aes(color=species,shape=island))
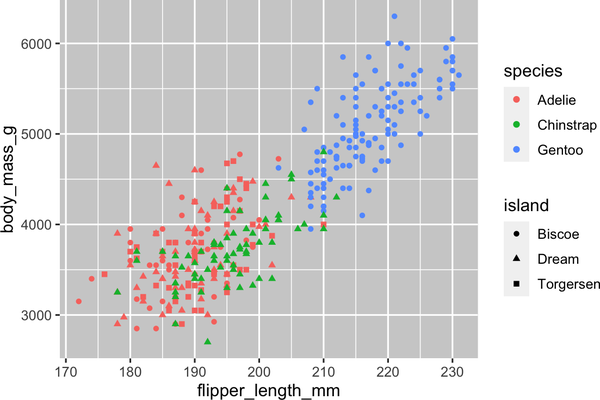
Sin embargo, añadir demasiados mapeados estéticos a un gráfico lo desordena y dificulta su comprensión. Otra opción, especialmente útil para las variables categóricas, es dividir tu gráfico en facetas, subparcelas que muestren cada una un subconjunto de datos.
Para facetar tu gráfico por una sola variable, utiliza facet_wrap(). El primer argumento de facet_wrap() es una fórmula3 que creas con ~ seguido del nombre de una variable. La variable que pases a facet_wrap() debe ser categórica.
ggplot(penguins,aes(x=flipper_length_mm,y=body_mass_g))+geom_point(aes(color=species,shape=species))+facet_wrap(~island)
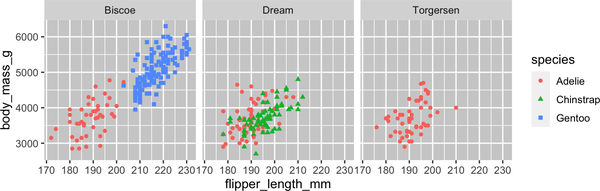
En el Capítulo 9 conocerás muchas otras geomáticas para visualizar distribuciones de variables y relaciones entre ellas.
Ejercicios
El marco de datos
mpgque se incluye con el paquete ggplot2 contiene 234 observaciones recogidas por la Agencia de Protección Medioambiental de EE.UU. sobre 38 modelos de coche. ¿Qué variables dempgson categóricas? ¿Qué variables son numéricas? (Sugerencia: teclea?mpgpara leer la documentación del conjunto de datos). ¿Cómo puedes ver esta información cuando ejecutesmpg?Haz un gráfico de dispersión de
hwyfrente adisplutilizando el marco de datosmpg. A continuación, asigna una tercera variable numérica acolor, luego asize, luego tanto acolorcomo asize, y luego ashape. ¿Cómo se comportan estas estéticas de forma diferente para las variables categóricas frente a las numéricas?En el diagrama de dispersión de
hwyfrente adispl, ¿qué ocurre si asignas una tercera variable alinewidth?¿Qué ocurre si asignas la misma variable a varias estéticas?
Haz un diagrama de dispersión de
bill_depth_mmfrente abill_length_mmy colorea los puntos porspecies. ¿Qué revela la coloración por especies sobre la relación entre estas dos variables? ¿Y el facetado por especies?-
¿Por qué lo siguiente produce dos leyendas separadas? ¿Cómo lo arreglarías para combinar las dos leyendas?
ggplot(data=penguins,mapping=aes(x=bill_length_mm,y=bill_depth_mm,color=species,shape=species))+geom_point()+labs(color="Species") -
Crea los dos gráficos de barras apiladas siguientes. ¿Qué pregunta puedes responder con el primero? ¿Qué pregunta puedes responder con el segundo?
ggplot(penguins,aes(x=island,fill=species))+geom_bar(position="fill")ggplot(penguins,aes(x=species,fill=island))+geom_bar(position="fill")
Guardar tus parcelas
Una vez que hayas hecho un trazado, puede que quieras sacarlo de R guardándolo como una imagen que puedas utilizar en otro sitio. Esa es la función de ggsave()que guardará en disco el último gráfico creado:
ggplot(penguins,aes(x=flipper_length_mm,y=body_mass_g))+geom_point()ggsave(filename="penguin-plot.png")
Esto guardará tu trama en tu directorio de trabajo, un concepto sobre el que aprenderás más en el Capítulo 6.
Si no especificas width y height, se tomarán de las dimensiones del dispositivo de trazado actual. Para un código reproducible, querrás especificarlas. Puedes obtener más información sobre ggsave() en la documentación.
En general, sin embargo, te recomendamos que montes tus informes finales utilizando Quarto, un sistema de autoría reproducible que te permite intercalar tu código y tu prosa e incluir automáticamente tus tramas en tus escritos. Obtendrás más información sobre Quarto en el Capítulo 28.
Ejercicios
-
Ejecuta las siguientes líneas de código. ¿Cuál de los dos gráficos se guarda como
mpg-plot.png? ¿Por qué?ggplot(mpg,aes(x=class))+geom_bar()ggplot(mpg,aes(x=cty,y=hwy))+geom_point()ggsave("mpg-plot.png") ¿Qué tienes que cambiar en el código anterior para guardar el gráfico como PDF en lugar de como PNG? ¿Cómo podrías averiguar qué tipos de archivos de imagen funcionarían en
ggsave()?
Problemas comunes
Cuando empieces a ejecutar código R, es probable que te encuentres con problemas. No te preocupes: le ocurre a todo el mundo. Todos llevamos años escribiendo código R, ¡pero cada día seguimos escribiendo código que no funciona al primer intento!
Empieza por comparar cuidadosamente el código que estás ejecutando con el código del libro. R es extremadamente quisquilloso, y un carácter mal colocado puede marcar la diferencia. Asegúrate de que cada ( se empareja con un ) y cada " se empareja con otro ". A veces ejecutarás el código y no ocurrirá nada. Comprueba la parte izquierda de tu consola: si es un +, significa que R no cree que hayas escrito una expresión completa y está esperando a que la termines. En este caso, suele ser fácil volver a empezar desde cero pulsando Escape para abortar el procesamiento del comando actual.
Un problema habitual al crear gráficos ggplot2 es colocar el + en el lugar equivocado: tiene que ir al final de la línea, no al principio. En otras palabras, asegúrate de que no has escrito accidentalmente un código como éste:
ggplot(data=mpg)+geom_point(mapping=aes(x=displ,y=hwy))
Si sigues atascado, prueba la ayuda. Puedes obtener ayuda sobre cualquier función de R ejecutando ?function_name en la consola o resaltando el nombre de la función y pulsando F1 en RStudio. No te preocupes si la ayuda no te parece muy útil; en lugar de eso, salta a los ejemplos y busca el código que coincida con lo que intentas hacer.
Si eso no te ayuda, lee atentamente el mensaje de error. ¡A veces la respuesta estará enterrada allí! Pero cuando eres nuevo en R, aunque la respuesta esté en el mensaje de error, puede que aún no sepas cómo entenderla. Otra gran herramienta es Google: intenta buscar en Google el mensaje de error, ya que es probable que alguien haya tenido el mismo problema y haya obtenido ayuda en línea.
Resumen
En este capítulo, has aprendido los fundamentos de la visualización de datos con ggplot2. Empezamos con la idea básica que sustenta ggplot2: una visualización es un mapeo desde variables de tus datos a propiedades estéticas como posición, color, tamaño y forma. A continuación, aprendiste a aumentar la complejidad y mejorar la presentación de tus gráficos capa a capa. También aprendiste sobre los trazados de uso común para visualizar la distribución de una sola variable, así como para visualizar las relaciones entre dos o más variables, aprovechando mapeados estéticos adicionales y/o dividiendo tu trazado en pequeños múltiplos mediante facetas.
Utilizaremos visualizaciones una y otra vez a lo largo de este libro, introduciendo nuevas técnicas a medida que las necesitemos, además de profundizar en la creación de visualizaciones con ggplot2 en los capítulos 9 a 11.
Ahora que ya conoces los fundamentos de la visualización, en el siguiente capítulo cambiaremos un poco de marcha y te daremos algunos consejos prácticos sobre el flujo de trabajo. Intercalamos consejos de flujo de trabajo con herramientas de ciencia de datos en toda esta parte del libro, porque te ayudarán a mantenerte organizado mientras escribes cantidades cada vez mayores de código R.
1 Puedes eliminar ese mensaje y forzar que la resolución de conflictos se produzca bajo demanda utilizando el paquete en conflicto, que se vuelve más importante a medida que cargas más paquetes. Puedes obtener más información sobre conflicted en el sitio web de paquetes.
2 Horst AM, Hill AP, Gorman KB (2020). pingüinos palmer: Datos de pingüinos del Archipiélago Palmer (Antártida). Paquete R versión 0.1.0. https://oreil.ly/ncwc5. doi: 10.5281/zenodo.3960218.
3 Aquí "fórmula" es el nombre de lo creado por ~, no un sinónimo de "ecuación".
Get R para la Ciencia de Datos, 2ª Edición now with the O’Reilly learning platform.
O’Reilly members experience books, live events, courses curated by job role, and more from O’Reilly and nearly 200 top publishers.

