Capítulo 13. Sitios de Restricción de Ubicación: Utilizar, probar y compartir código
Este trabajo se ha traducido utilizando IA. Agradecemos tus opiniones y comentarios: translation-feedback@oreilly.com
Una secuencia palindrómica en el ADN es aquella en la que la secuencia de pares de bases 5' a 3' es idéntica en ambas cadenas. Por ejemplo, la Figura 13-1 muestra que el complemento inverso de la secuencia de ADN GCATGC es la propia secuencia.
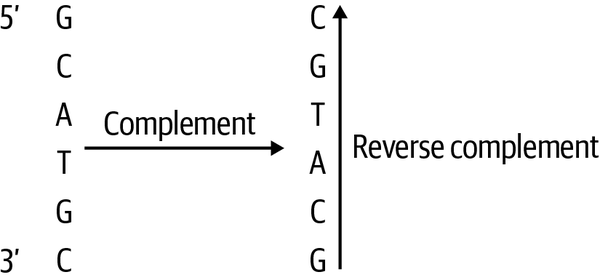
Figura 13-1. Un palíndromo inverso es igual a su complemento inverso
Puedo verificarlo en código:
>>> from Bio import Seq >>> seq = 'GCATGC' >>> Seq.reverse_complement(seq) == seq True
Como se describe en el reto REVP de Rosalind, las enzimas de restricción reconocen y cortan dentro de secuencias palindrómicas específicas de ADN conocidas como sitios de restricción. Suelen tener una longitud de entre 4 y 12 nucleótidos. El objetivo de este ejercicio es encontrar las ubicaciones en una secuencia de ADN de cada enzima de restricción putativa. El código para resolver este problema podría ser enormemente complicado, pero una comprensión clara de algunas técnicas de programación funcional ayuda a crear una solución breve y elegante. Exploraré map(), zip() y enumerate(), así como muchas funciones pequeñas y probadas.
Aprenderás:
-
Cómo encontrar un palíndromo inverso
-
Cómo crear módulos para compartir funciones comunes
-
Acerca de la variable ...
Get Dominar Python para Bioinformática now with the O’Reilly learning platform.
O’Reilly members experience books, live events, courses curated by job role, and more from O’Reilly and nearly 200 top publishers.

