Capítulo 1. Frecuencia de los tetranucleótidos: Contando cosas
Este trabajo se ha traducido utilizando IA. Agradecemos tus opiniones y comentarios: translation-feedback@oreilly.com
Contar las bases en el ADN es quizás el "¡Hola, mundo!" de la bioinformática. El reto Rosalind DNA describe un programa que tomará una secuencia de ADN e imprimirá un recuento de cuántos As, Cs, Gsy Tsse encuentran. Hay sorprendentemente muchas formas de contar cosas en Python, y exploraré lo que el lenguaje puede ofrecer. También demostraré cómo escribir un programa bien estructurado y documentado que valide sus argumentos, así como cómo escribir y ejecutar pruebas para garantizar que el programa funciona correctamente.
En este capítulo aprenderás
-
Cómo iniciar un nuevo programa utilizando
new.py -
Cómo definir y validar los argumentos de la línea de comandos utilizando
argparse -
Cómo ejecutar un conjunto de pruebas con
pytest -
Cómo iterar los caracteres de una cadena
-
Formas de contar los elementos de una colección
-
Cómo crear un árbol de decisión utilizando las declaraciones
if/elif -
Cómo formatear cadenas
Cómo empezar
Antes de empezar, asegúrate de haber leído "Cómo obtener el código y las pruebas" en el Prefacio. Una vez que tengas una copia local del repositorio de código, cámbiate al directorio 01_dna:
$ cd 01_dna
Aquí encontrarás varios programas solution*.py junto con pruebas y datos de entrada que puedes utilizar para ver si los programas funcionan correctamente. Para hacerte una idea de cómo debería funcionar tu programa, empieza copiando la primera solución en un programa llamado dna.py:
$ cp solution1_iter.py dna.py
Ahora ejecuta el programa sin argumentos, o con las banderas -h o --help. Imprimirá la documentación de uso (ten en cuenta que uso es la primera palabra de la salida):
$ ./dna.py usage: dna.py [-h] DNA dna.py: error: the following arguments are required: DNA
Si obtienes un error como "permiso denegado", puede que tengas que ejecutar chmod +x dna.py para cambiar el modo del programa añadiendo el bit ejecutable.
Éste es uno de los primeros elementos de la reproducibilidad.Los programas deben proporcionar documentación sobre su funcionamiento.Aunque es habitual tener algo como un archivo README o incluso un artículo para describir un programa, el propio programa debe proporcionar documentación sobre sus parámetros y salidas.
Te mostraré cómo utilizar el módulo argparse para definir y validar los argumentos, así como para generar la documentación, lo que significa que no hay posibilidad de que la declaración de uso generada por el programa pueda ser incorrecta. Contrasta esto con la forma en que los archivos README y los registros de cambios y similares pueden desincronizarse rápidamente con el desarrollo de un programa, y espero que aprecies que este tipo de documentación es bastante eficaz.
Puedes ver en la línea de uso que el programa espera algo como DNA como argumento, así que vamos a darle una secuencia.Como se describe en la página de Rosalind, el programa imprime los recuentos de cada una de las bases A, C, G y T, en ese orden y separadas por un solo espacio cada una:
$ ./dna.py ACCGGGTTTT 1 2 3 4
Cuando vayas a resolver un reto en el sitio web Rosalind.info, la entrada para tu programa se proporcionará como un archivo descargado; por lo tanto, escribiré el programa para que también lea el contenido de un archivo. Puedo utilizar el comando cat (para concatenar) para imprimir el contenido de uno de los archivos del directorio tests/entradas:
$ cat tests/inputs/input2.txt AGCTTTTCATTCTGACTGCAACGGGCAATATGTCTCTGTGTGGATTAAAAAAAGAGTGTCTGATAGCAGC
Ésta es la misma secuencia que se muestra en el ejemplo de la página web. Por consiguiente, sé que la salida del programa debería ser ésta:
$ ./dna.py tests/inputs/input2.txt 20 12 17 21
A lo largo del libro, utilizaré la herramienta pytest para ejecutar las pruebas que garantizan que los programas funcionan como se espera de ellos. Cuando ejecute el comando pytestbuscará recursivamente en el directorio actual pruebas y funciones que parezcan pruebas. Ten en cuenta que puede que necesites ejecutar python3 -m pytest o pytest.exe si estás en Windows. Ejecútalo ahora, y deberías ver algo como lo siguiente para indicar que el programa supera las cuatro pruebas que se encuentran en el archivo tests/dna_test.py:
$ pytest =========================== test session starts =========================== ... collected 4 items tests/dna_test.py .... [100%] ============================ 4 passed in 0.41s ============================
Un elemento clave para probar el software es ejecutar el programa con entradas conocidas y verificar que produce la salida correcta.Aunque pueda parecer una idea obvia, he tenido que oponerme a esquemas de "prueba" que simplemente ejecutaban programas pero nunca verificaban que se comportaran correctamente.
Crear el programa con nuevo.py
Si has copiado una de las soluciones, como se indica en el apartado anterior, elimina ese programa para poder empezar de cero:
$ rm dna.py
Sin mirar todavía mis soluciones, quiero que intentes resolver este problema. Si crees que tienes toda la información que necesitas, no dudes en lanzarte y escribir tu propia versión de dna.py, utilizando pytest para ejecutar las pruebas proporcionadas. Sigue leyendo si quieres seguir conmigo paso a paso para aprender a escribir el programa y ejecutar las pruebas.
Todos los programas de este libro aceptarán algún(os) argumento(s) de la línea de comandos y crearán alguna salida, como texto en la línea de comandos o nuevos archivos. Siempre utilizaré el programa new.py descrito en el Prefacio para empezar, pero esto no es un requisito. Puedes escribir tus programas como quieras, empezando desde el punto que quieras, pero se espera que tus programas tengan las mismas características, como generar declaraciones de uso y validar adecuadamente los argumentos.
Crea tu programa dna.py en el directorio 01_dna, ya que éste contiene los archivos de prueba del programa. Así es como iniciaré el programa dna.py. El argumento --purpose se utilizará en la documentación del programa:
$ new.py --purpose 'Tetranucleotide frequency' dna.py Done, see new script "dna.py."
Si ejecutas el nuevo programa dna.py, verás que define muchos tipos diferentes de argumentos comunes a los programas de línea de comandos:
$ ./dna.py --help usage: dna.py [-h] [-a str] [-i int] [-f FILE] [-o] str Tetranucleotide frequencypositional arguments: str A positional argument
optional arguments: -h, --help show this help message and exit
-a str, --arg str A named string argument (default: )
-i int, --int int A named integer argument (default: 0)
-f FILE, --file FILE A readable file (default: None)
-o, --on A boolean flag (default: False)

El
--purposedenew.pyse utiliza aquí para describir el programa.

Las banderas
-hy--helpson añadidas automáticamente porargparsey activarán el uso.
Se trata de una opción con nombre, con nombres cortos (
-a) y largos (--arg) para un valor de cadena.
Se trata de una opción con nombres cortos (
-i) y largos (--int) para un valor entero.
Se trata de una opción con nombre, con nombres cortos (
-f) y largos (--file) para un argumento de archivo .
Se trata de una bandera booleana que será
Truecuando-oo--onestén presentes yFalsecuando estén ausentes.
Este programa sólo necesita el argumento posicional str, y puedes utilizar DNA para el valor metavar para dar alguna indicación al usuario sobre el significado del argumento. Elimina todos los demás parámetros. Ten en cuenta que nunca debes definir los indicadores -h y --help, ya que argparse los utiliza internamente para responder a las solicitudes de uso. Comprueba si puedes modificar tu programa hasta que produzca el uso que se muestra a continuación (si todavía no puedes producir el uso, no te preocupes, te lo mostraré en la siguiente sección):
$ ./dna.py -h usage: dna.py [-h] DNA Tetranucleotide frequency positional arguments: DNA Input DNA sequence optional arguments: -h, --help show this help message and exit
Si consigues hacerlo funcionar, me gustaría señalar que este programa aceptará exactamente un argumento posicional. Si intentas ejecutarlo con cualquier otro número de argumentos, el programa se detendrá inmediatamente e imprimirá un mensaje de error:
$ ./dna.py AACC GGTT usage: dna.py [-h] DNA dna.py: error: unrecognized arguments: GGTT
Del mismo modo, el programa rechazará cualquier bandera u opción desconocida. Con muy pocas líneas de código, has construido un programa documentado que valida los argumentos del programa.Ése es un paso muy básico e importante hacia la reproducibilidad.
Utilizar argparse
El programa creado por new.py utiliza el módulo argparse para definir los parámetros del programa, validar que los argumentos son correctos y crear la documentación de uso para el usuario. El módulo argparse es un módulo estándar de Python, lo que significa que siempre está presente.Otros módulos también pueden hacer estas cosas, y eres libre de utilizar el método que quieras para manejar este aspecto de tu programa. Sólo asegúrate de que tus programas pueden superar las pruebas.
Escribí una versión de new.py para Tiny Python Projects que puedes encontrar en el directorio bin del repositorio GitHub de ese libro.Esa versión es algo más sencilla que la versión que quiero que utilices. Empezaré mostrándote una versión de dna.py creada utilizando esta versión anterior de new.py:
#!/usr/bin/env python3""" Tetranucleotide frequency """
import argparse
# -------------------------------------------------- def get_args():
""" Get command-line arguments """
parser = argparse.ArgumentParser(
description='Tetranucleotide frequency', formatter_class=argparse.ArgumentDefaultsHelpFormatter) parser.add_argument('dna', metavar='DNA', help='Input DNA sequence')
return parser.parse_args()
# -------------------------------------------------- def main():
""" Make a jazz noise here """ args = get_args()
print(args.dna)
# -------------------------------------------------- if __name__ == '__main__':
main()

El coloquial shebang (
#!) indica al sistema operativo que utilice el comandoenv(entorno) para encontrarpython3y ejecutar el resto del programa.
Es una docstring (cadena de documentación) del programa o módulo en su conjunto.

Importo el módulo
argparsepara manejar los argumentos de la línea de comandos.
Siempre defino una función
get_args()para gestionar el códigoargparse.
Esto es un docstring para una función.

El objeto
parserse utiliza para definir los parámetros del programa.
Defino un argumento
dna, que será posicional porque el nombrednano empieza por guión. Elmetavares una breve descripción del argumento que aparecerá en el uso abreviado. No se necesita ningún otro argumento.
La función devuelve los resultados del análisis sintáctico de los argumentos. Las banderas de ayuda o cualquier problema con los argumentos harán que
argparseimprima una declaración de uso/mensajes de error y salga del programa.
Todos los programas del libro empezarán siempre en la función
main().
El primer paso en
main()será siempre llamar aget_args(). Si esta llamada tiene éxito, entonces los argumentos deben haber sido válidos.
El valor
DNAestá disponible en el atributoargs.dna, ya que es el nombre del argumento.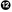
Se trata de un modismo habitual en los programas Python para detectar cuándo se está ejecutando el programa (en lugar de ser importado) y ejecutar la función
main().
La línea shebang es utilizada por el shell de Unix cuando se invoca al programa como un programa, como ./dna.py. No funciona en Windows, donde se requiere ejecutar python.exe dna.py para ejecutar el programa.
Aunque este código funciona de forma totalmente adecuada, el valor devuelto por get_args() es un objeto argparse.Namespace que se genera dinámicamente cuando se ejecuta el programa.Es decir, estoy utilizando código como parser.add_argument() para modificar la estructura de este objeto en tiempo de ejecución, por lo que Python es incapaz de saber positivamente en tiempo de compilación qué atributos estarán disponibles en los argumentos analizados o cuáles serían sus tipos. Aunque puede resultarte obvio que sólo puede haber un único argumento de cadena obligatorio, no hay suficiente información en el código para que Python pueda discernirlo.
Compilar un programa es convertirlo en el código máquina que un ordenador puede ejecutar. Algunos lenguajes, como el C, deben compilarse por separado antes de poder ejecutarse. Los programas Python suelen compilarse y ejecutarse en un solo paso, pero sigue habiendo una fase de compilación. Algunos errores pueden detectarse en la compilación, y otros no aparecen hasta el momento de la ejecución. Por ejemplo, los errores de sintaxis impedirán la compilación. Es preferible tener errores en tiempo de compilación que en tiempo de ejecución.
Para ver por qué esto puede ser un problema, alteraré la función main() para introducir un error de tipo. Es decir, utilizaré mal intencionadamente el tipo del valor args.dna. A menos que se indique lo contrario, todos los valores de los argumentos devueltos desde la línea de comandos por argparse son cadenas. Si intento dividir la cadena args.dna por el valor entero 2, Python lanzará una excepción y bloqueará el programa en tiempo de ejecución:
def main():
args = get_args()
print(args.dna / 2) 
Si ejecuto el programa, se bloquea como era de esperar:
$ ./dna.py ACGT
Traceback (most recent call last):
File "./dna.py", line 30, in <module>
main()
File "./dna.py", line 25, in main
print(args.dna / 2)
TypeError: unsupported operand type(s) for /: 'str' and 'int'
Nuestros grandes cerebros blandengues saben que se trata de un error inevitable a punto de producirse, pero Python no ve el problema. Lo que necesito es una definición estática de los argumentos que no pueda modificarse al ejecutar el programa. Sigue leyendo para ver cómo las anotaciones de tipo y otras herramientas pueden detectar este tipo de errores.
Herramientas para encontrar errores en el código
El objetivo aquí es escribir programas correctos y reproducibles en Python.¿Existen formas de detectar y evitar problemas como el uso incorrecto de una cadena en una operación numérica?El intérprete python3 no encontró ningún problema que me impidiera ejecutar el código.
Es decir, el programa es sintácticamente correcto, por lo que el código de la sección anterior produce un error en tiempo de ejecución porque el error sólo se produce cuando ejecuto el programa.Hace años trabajé en un grupo en el que bromeábamos: "¡Si compila, envíalo!" Éste es claramente un enfoque miope a la hora de codificar en Python.
Puedo utilizar herramientas como linters y comprobadores de tipos para encontrar algunos tipos de problemas en el código.Los linters son herramientas que comprueban el estilo del programa y muchos tipos de errores más allá de la mala sintaxis. La herramientapylint es un popular linter de Python que utilizo casi todos los días.¿Puede encontrar este problema? Aparentemente no, ya que da el mayor de los pulgares hacia arriba:
$ pylint dna.py ------------------------------------------------------------------- Your code has been rated at 10.00/10 (previous run: 9.78/10, +0.22)
La herramienta flake8 es otro linter que suelo utilizar en combinación con pylint, ya que informará de distintos tipos de errores.Cuando ejecuto flake8 dna.py, no obtengo ninguna salida, lo que significa que no ha encontrado errores de los que informar.
La herramienta mypy es un comprobador de tipos estático para Python, , lo que significa que está diseñado para encontrar tipos mal utilizados, como intentar dividir una cadena por un número. Ni pylint ni flake8 están diseñados para detectar errores de tipo, por lo que no puedo sorprenderme legítimamente de que pasaran por alto el error. Entonces, ¿qué tiene que decir mypy?
$ mypy dna.py Success: no issues found in 1 source file
Bueno, eso es un poco decepcionante; sin embargo, debes entender que mypy no informa de un problema porque no hay información de tipo. Es decir, mypy no tiene información para decir que dividir args.dna por 2 es incorrecto. Lo arreglaré en breve.
Introducción a las tuplas con nombre
Para evitar los problemas con los objetos generados dinámicamente, todos los programas de este libro utilizarán una estructura de datos de tuplas con nombre para definir estáticamente los argumentos de get_args().Las tuplas son esencialmente listas inmutables, y se utilizan a menudo para representar estructuras de datos de tipo registro en Python. Hay mucho que desentrañar con todo esto, así que volvamos a las listas.
Para empezar, las listas son secuencias ordenadas de elementos. Los elementos pueden ser heterogéneos; en teoría, esto significa que todos los elementos pueden ser de distintos tipos, pero en la práctica, mezclar tipos suele ser una mala idea. Utilizaré el REPL de python3 para demostrar algunos aspectos de las listas.Te recomiendo que utilices help(list) para leer la documentación.
Utiliza corchetes vacíos ([]) para crear una lista vacía que contendrá algunas secuencias:
>>> seqs = []
La función list() también creará una nueva lista vacía:
>>> seqs = list()
Comprueba que se trata de una lista utilizando la función type() para devolver el tipo de la variable:
>>> type(seqs) <class 'list'>
Las listas tienen métodos que añaden valores al final de la lista, como list.append() para añadir un valor:
>>> seqs.append('ACT')
>>> seqs
['ACT']
y list.extend() para añadir varios valores:
>>> seqs.extend(['GCA', 'TTT']) >>> seqs ['ACT', 'GCA', 'TTT']
Si escribes la variable sola en el REPL, se evaluará y se convertirá en una representación textual:
>>> seqs ['ACT', 'GCA', 'TTT']
Esto es básicamente lo mismo que ocurre cuando print() una variable:
>>> print(seqs) ['ACT', 'GCA', 'TTT']
Puedes modificar cualquiera de los valores in situ utilizando el índice. Recuerda que toda indexación en Python está basada en 0, por lo que 0 es el primer elemento.Cambia la primera secuencia para que sea TCA:
>>> seqs[0] = 'TCA'
Comprueba que se ha cambiado:
>>> seqs ['TCA', 'GCA', 'TTT']
Al igual que las listas, las tuplas son secuencias ordenadas de objetos posiblemente heterogéneos. Siempre que pongas comas entre los elementos de una serie, estarás creando una tupla:
>>> seqs = 'TCA', 'GCA', 'TTT' >>> type(seqs) <class 'tuple'>
Es típico poner paréntesis alrededor de los valores de las tuplas para hacer esto más explícito:
>>> seqs = ('TCA', 'GCA', 'TTT')
>>> type(seqs)
<class 'tuple'>
A diferencia de las listas, las tuplas no pueden modificarse una vez creadas. Si lees help(tuple), verás que una tupla es una secuencia inmutable incorporada, por lo que no puedo añadir valores:
>>> seqs.append('GGT')
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
AttributeError: 'tuple' object has no attribute 'append'
o modificar los valores existentes:
>>> seqs[0] = 'TCA' Traceback (most recent call last): File "<stdin>", line 1, in <module> TypeError: 'tuple' object does not support item assignment
Es bastante habitual en Python utilizar tuplas para representar registros.Por ejemplo, podría representar un Sequence que tuviera un ID único y una cadena de bases:
>>> seq = ('CAM_0231669729', 'GTGTTTATTCAATGCTAG')
Aunque es posible utilizar la indexación para obtener los valores de una tupla igual que con las listas, eso es incómodo y propenso a errores.Las tuplas con nombre me permiten asignar nombres a los campos, lo que hace que su uso sea más ergonómico. Para utilizar tuplas con nombre, puedo importar la función namedtuple() del módulo collections:
>>> from collections import namedtuple
Como se muestra en la Figura 1-2, utilizo la función namedtuple() para crear la idea de un Sequence que tiene campos para el id y el seq:
>>> Sequence = namedtuple('Sequence', ['id', 'seq'])
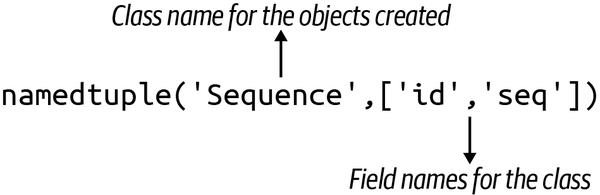
Figura 1-2. La función namedtuple() genera una forma de hacer objetos de la clase Sequence que tengan los campos id y seq
¿Qué es exactamente Sequence?
>>> type(Sequence) <class 'type'>
Acabo de crear un nuevo tipo. Podrías llamar fábrica a la función Sequence() porque es una función que se utiliza para generar nuevos objetos de la clase Sequence.Es una convención de nomenclatura común que estas funciones de fábrica y los nombres de clase se pongan TitleCased para diferenciarlos.
Igual que puedo utilizar la función list() para crear una nueva lista, puedo utilizar la función Sequence() para crear un nuevo objeto Sequence. Puedo pasar los valores id y seq posicionalmente para que coincidan con el orden en que están definidos en la clase:
>>> seq1 = Sequence('CAM_0231669729', 'GTGTTTATTCAATGCTAG')
>>> type(seq1)
<class '__main__.Sequence'>
O puedo utilizar los nombres de los campos y pasarlos como pares clave/valor en el orden que quiera:
>>> seq2 = Sequence(seq='GTGTTTATTCAATGCTAG', id='CAM_0231669729') >>> seq2 Sequence(id='CAM_0231669729', seq='GTGTTTATTCAATGCTAG')
Aunque es posible utilizar índices para acceder al ID y a la secuencia:
>>> 'ID = ' + seq1[0] 'ID = CAM_0231669729' >>> 'seq = ' + seq1[1] 'seq = GTGTTTATTCAATGCTAG'
...el objetivo de las tuplas con nombre es utilizar los nombres de los campos:
>>> 'ID = ' + seq1.id 'ID = CAM_0231669729' >>> 'seq = ' + seq1.seq 'seq = GTGTTTATTCAATGCTAG'
Los valores del registro permanecen inmutables:
>>> seq1.id = 'XXX' Traceback (most recent call last): File "<stdin>", line 1, in <module> AttributeError: can't set attribute
A menudo quiero una garantía de que un valor no puede cambiarse accidentalmente en mi código. Python no tiene una forma de declarar que una variable es constante o inmutable. Las tuplas son inmutables por defecto, y creo que tiene sentido representar los argumentos de un programa utilizando una estructura de datos que no pueda alterarse. Las entradas son sacrosantas y no deberían modificarse (casi) nunca.
Añadir tipos a tuplas con nombre
Por muy bonito que sea namedtuple(), puedo mejorarlo aún más importando la clase NamedTuple del módulo typing para utilizarla como clase base de Sequence. Además, puedo asignar tipos a los campos utilizando esta sintaxis. Observa la necesidad de utilizar una línea vacía en el REPL para indicar que el bloque está completo:
>>> from typing import NamedTuple >>> class Sequence(NamedTuple): ... id: str ... seq: str ...
Los ... que ves son continuaciones de línea. El REPL está mostrando que lo que se ha introducido hasta ahora no es una expresión completa. Necesitas introducir una línea en blanco para que el REPL sepa que has terminado con el bloque de código.
Al igual que el método namedtuple(), Sequence es un tipo nuevo:
>>> type(Sequence) <class 'type'>
El código para instanciar un nuevo objeto Sequence es el mismo:
>>> seq3 = Sequence('CAM_0231669729', 'GTGTTTATTCAATGCTAG')
>>> type(seq3)
<class '__main__.Sequence'>
Todavía puedo acceder a los campos por los nombres:
>>> seq3.id, seq3.seq
('CAM_0231669729', 'GTGTTTATTCAATGCTAG')
Como he definido que ambos campos tienen tipos str, podrías suponer que esto no funcionaría:
>>> seq4 = Sequence(id='CAM_0231669729', seq=3.14)
Siento decirte que el propio Python ignora la información del tipo.Puedes ver que el campo seq que esperaba que fuera un str es en realidad un float:
>>> seq4 Sequence(id='CAM_0231669729', seq=3.14) >>> type(seq4.seq) <class 'float'>
Entonces, ¿en qué nos ayuda esto? A mí no me ayuda en la REPL, pero añadir tipos a mi código fuente permitirá que herramientas de comprobación de tipos como mypy encuentren esos errores.
Representar los argumentos con una tupla con nombre
Quiero que la estructura de datos que representa los argumentos del programa incluya información de tipo. Al igual que con la clase Sequence, puedo definir una clase derivada del tipo NamedTuple en la que puedo definir estáticamente la estructura de datos con tipos. Me gusta llamar a esta clase Args, pero puedes llamarla como quieras. Sé que esto probablemente parezca clavar un clavo de remate con un mazo, pero confía en mí, este tipo de detalles valdrán la pena en el futuro.
La última versión de new.py utiliza la clase NamedTuple del módulo typing. He aquí cómo te sugiero que definas y representes los argumentos:
#!/usr/bin/env python3 """Tetranucleotide frequency""" import argparse from typing import NamedTupleclass Args(NamedTuple):
""" Command-line arguments """ dna: str
# -------------------------------------------------- def get_args() -> Args:
""" Get command-line arguments """ parser = argparse.ArgumentParser( description='Tetranucleotide frequency', formatter_class=argparse.ArgumentDefaultsHelpFormatter) parser.add_argument('dna', metavar='DNA', help='Input DNA sequence') args = parser.parse_args()
return Args(args.dna)
# -------------------------------------------------- def main() -> None:
""" Make a jazz noise here """ args = get_args() print(args.dna / 2)
# -------------------------------------------------- if __name__ == '__main__': main()

Importa la clase
NamedTupledel módulotyping.
Define un
classpara los argumentos que se base en la claseNamedTuple. Consulta la nota siguiente.
La clase tiene un único campo llamado
dnaque tiene el tipostr.
La anotación de tipo de la función
get_args()muestra que devuelve un objeto del tipoArgs.
Analiza los argumentos como antes.

Devuelve un nuevo objeto
Argsque contiene el valor único deargs.dna.
La función
main()no tiene ninguna declaraciónreturn, por lo que devuelve el valor por defectoNone.
Este es el error de tipo del programa anterior.
Si ejecutas pylint en este programa, puedes encontrarte con los errores "Heredando NamedTuple, que no es una clase. (heredar-no-clase)" y "Demasiados pocos métodos públicos (0/2) (too-few-public-methods)". Puedes desactivar estas advertencias añadiendo "inherit-non-class" y "too-few-public-methods" a la sección "disable" de tu archivo pylintrc, o utiliza el archivo pylintrc incluido en la raíz del repositorio de GitHub.
Si ejecutas este programa, verás que sigue creando la misma excepción no capturada. Tanto flake8 como pylint seguirán informando de que el programa parece correcto, pero mira lo que me dice ahora mypy:
$ mypy dna.py
dna.py:32: error: Unsupported operand types for / ("str" and "int")
Found 1 error in 1 file (checked 1 source file)
El mensaje de error muestra que hay un problema en la línea 32 con los operandos, que son los argumentos del operador de división (/). Estoy mezclando valores de cadena y enteros. Sin las anotaciones de tipo, mypy sería incapaz de encontrar un error. Sin esta advertencia de mypy, tendría que ejecutar mi programa para encontrarlo, asegurándome de ejercitar la rama de código que contiene el error.
En este caso, todo es bastante obvio y trivial, pero en un programa mucho más grande con cientos o miles de líneas de código (LOC) con muchas funciones y ramas lógicas (como if/else), podría no tropezar con este error. Confío en tipos y programas como mypy (y pylint y flake8 y así sucesivamente) para corregir este tipo de errores en lugar de confiar únicamente en las pruebas, o peor aún, esperar a que los usuarios informen de los errores.
Lectura de entradas de la línea de comandos o de un archivo
Cuando intentes probar que tu programa funciona en el sitio web Rosalind.info, descargarás un archivo de datos que contiene la entrada de tu programa. Normalmente, estos datos serán mucho mayores que los datos de ejemplo descritos en el problema. Por ejemplo, la cadena de ADN de ejemplo para este problema tiene 70 bases de longitud, pero la que descargué para uno de mis intentos tenía 910 bases.
Hagamos que el programa lea la entrada tanto de la línea de comandos como de un archivo de texto, para que no tengas que copiar y pegar el contenido de un archivo descargado. Éste es un patrón habitual que utilizo, y prefiero manejar esta opción dentro de la función get_args(), ya que se refiere al procesamiento de los argumentos de la línea de comandos.
Primero, corrige el programa para que imprima el valor args.dna sin la división:
def main() -> None:
args = get_args()
print(args.dna) 
Comprueba que funciona:
$ ./dna.py ACGT ACGT
Para la siguiente parte, tienes que introducir el módulo os para que interactúe con tu sistema operativo. Añade import os a las otras declaraciones import de la parte superior y, a continuación, añade estas dos líneas a tu función get_args():
def get_args() -> Args:
""" Get command-line arguments """
parser = argparse.ArgumentParser(
description='Tetranucleotide frequency',
formatter_class=argparse.ArgumentDefaultsHelpFormatter)
parser.add_argument('dna', metavar='DNA', help='Input DNA sequence')
args = parser.parse_args()
if os.path.isfile(args.dna):  args.dna = open(args.dna).read().rstrip()
args.dna = open(args.dna).read().rstrip()  return Args(args.dna)
return Args(args.dna)

Comprueba si el valor
dnaes un archivo.
Llama a
open()para abrir un manejador de archivo, luego encadena el métodofh.read()para devolver una cadena, luego encadena el métodostr.rstrip()para eliminar los espacios en blanco finales.
La función fh.read() leerá un archivo entero en una variable. En este caso, el archivo de entrada es pequeño y, por lo tanto, esto debería ir bien, pero es muy común en bioinformática procesar archivos que tienen un tamaño de gigabytes. Utilizar read() en un archivo grande podría bloquear tu programa o incluso todo tu ordenador. Más adelante te mostraré cómo leer un archivo línea por línea para evitarlo.
Ahora ejecuta tu programa con un valor de cadena para asegurarte de que funciona:
$ ./dna.py ACGT ACGT
y utiliza un archivo de texto como argumento:
$ ./dna.py tests/inputs/input2.txt AGCTTTTCATTCTGACTGCAACGGGCAATATGTCTCTGTGTGGATTAAAAAAAGAGTGTCTGATAGCAGC
Ahora tienes un programa flexible que lee la entrada de dos fuentes. Ejecuta mypy dna.py para asegurarte de que no hay problemas.
Probar tu programa
Sabes por la descripción de Rosalind que, dada la entrada ACGT, el programa debería imprimir 1 1 1 1, ya que ése es el número de As, Cs, Gsy Ts, respectivamente. En el directorio 01_dna/tests, hay un archivo llamado dna_test.py que contiene pruebas para el programa dna.py. Escribí estas pruebas para ti, para que veas cómo es desarrollar un programa utilizando un método que te diga con cierta certeza cuándo tu programa es correcto.
Escribí estas pruebas para ti, para que puedas ver cómo es desarrollar un programa utilizando un método que te diga con cierta certeza cuándo tu programa es correcto. Las pruebas son realmente básicas: dada una cadena de entrada, el programa debe imprimir los recuentos correctos de los cuatro nucleótidos. Cuando el programa informa de los números correctos, entonces funciona.
Dentro del directorio 01_dna, me gustaría que ejecutaras pytest (o python3 -m pytest o pytest.exe en Windows). El programa buscará recursivamente todos los archivos cuyos nombres empiecen por test_ o terminen por _test.py. A continuación, ejecutará cualquier función de estos archivos cuyos nombres empiecen por test_.
Cuando ejecutes pytestverás un montón de resultados, la mayoría de los cuales son pruebas que fallan. Para entender por qué fallan estas pruebas, echemos un vistazo al módulo tests/dna_test.py:
""" Tests for dna.py """import os
import platform
from subprocess import getstatusoutput
PRG = './dna.py'
RUN = f'python {PRG}' if platform.system() == 'Windows' else PRG
TEST1 = ('./tests/inputs/input1.txt', '1 2 3 4')
TEST2 = ('./tests/inputs/input2.txt', '20 12 17 21') TEST3 = ('./tests/inputs/input3.txt', '196 231 237 246')

Este es el docstring del módulo.

El módulo estándar
osinteractuará con el sistema operativo.
El módulo
platformse utiliza para determinar si se está ejecutando en Windows.
Del módulo
subprocessimporto una función para ejecutar el programadna.pyy capturar la salida y el estado.
Las siguientes líneas son variables globales del programa. Suelo evitar las globales excepto en mis pruebas. Aquí quiero definir algunos valores que utilizaré en las funciones. Me gusta utilizar MAYÚSCULAS_NOMBRE para resaltar la visibilidad global.

La variable
RUNdetermina cómo ejecutar el programadna.py. En Windows, se debe utilizar el comandopythonpara ejecutar un programa Python, pero en plataformas Unix, se puede ejecutar directamente el programadna.py.
Las variables
TEST*son tuplas que definen un archivo que contiene una cadena de ADN y la salida esperada del programa para esa cadena.
El módulo pytest ejecutará las funciones de prueba en el orden en que estén definidas en el archivo de prueba. Suelo estructurar mis pruebas de modo que progresen desde los casos más sencillos a los más complejos, por lo que normalmente no tiene sentido continuar después de un fallo. Por ejemplo, la primera prueba es siempre que el programa a probar exista. Si no existe, no tiene sentido ejecutar más pruebas. Te recomiendo que ejecutes pytest con la bandera -x para detenerte en la primera prueba que falle junto con la bandera -v para obtener una salida detallada.
Veamos la primera prueba. La función se llama test_exists() para que la encuentre pytest. En el cuerpo de la función, utilizo una o varias sentencias assert para comprobar si alguna condición es cierta.1 Aquí afirmo que el programa dna.py existe. Por eso tu programa debe existir en este directorio, de lo contrario no lo encontraría la prueba:
def test_exists():""" Program exists """ assert os.path.exists(PRG)

El nombre de la función debe empezar por
test_para que la encuentrepytest.
La función
os.path.exists()devuelveTruesi el argumento dado es un archivo. Si devuelveFalse, entonces la afirmación falla y esta prueba fallará.
La siguiente prueba que escribo es siempre para comprobar que el programa producirá una declaración de uso para las banderas -h y --help. La función subprocess.getstatusoutput() ejecutará el programa dna.py con las banderas de ayuda corta y larga. En cada caso, quiero ver que el programa imprime un texto que empieza por la palabra uso:. No es una prueba perfecta. No comprueba que la documentación sea exacta, sólo que parezca algo que podría ser una declaración de uso. No creo que todas las pruebas tengan que ser completamente exhaustivas. Aquí tienes la prueba:
def test_usage() -> None:
""" Prints usage """
for arg in ['-h', '--help']:  rv, out = getstatusoutput(f'{RUN} {arg}')
rv, out = getstatusoutput(f'{RUN} {arg}')  assert rv == 0
assert rv == 0  assert out.lower().startswith('usage:')
assert out.lower().startswith('usage:') 

Iterar sobre las banderas de ayuda corta y larga.

Ejecuta el programa con el argumento y captura el valor de retorno y la salida.

Comprueba que el programa informa de un valor de salida correcto de 0.

Afirma que la salida en minúsculas del programa comienza con el texto uso:.
Los programas de línea de comandos suelen indicar un error al sistema operativo devolviendo un valor distinto de cero. Si el programa se ejecuta correctamente, debería devolver un 0. A veces, ese valor distinto de cero puede correlacionarse con algún código de error interno, pero a menudo sólo significa que algo ha ido mal. Del mismo modo, los programas que escriba se esforzarán siempre por informar de 0 en caso de ejecución correcta y de algún valor distinto de cero cuando haya errores.
A continuación, quiero asegurarme de que el programa morirá cuando no se le den argumentos:
def test_dies_no_args() -> None:
""" Dies with no arguments """
rv, out = getstatusoutput(RUN)  assert rv != 0
assert rv != 0  assert out.lower().startswith('usage:')
assert out.lower().startswith('usage:') 

Captura el valor de retorno y la salida de la ejecución del programa sin argumentos.

Comprueba que el valor de retorno es un código de fallo distinto de cero.

Comprueba que la salida se parece a una declaración de uso.
En este punto de la prueba, sé que tengo un programa con el nombre correcto que puede ejecutarse para producir documentación. Esto significa que el programa es al menossintácticamente correcto, lo cual es un buen punto de partida para la prueba. Si tu programa tiene errores tipográficos, te verás obligado a corregirlos para llegar siquiera a este punto.
Ejecutar el programa para probar la salida
Ahora tengo que ver si el programa hace lo que se supone que debe hacer. Hay muchas formas de probar los programas, y a mí me gusta utilizar dos enfoques básicos que denomino de dentro a fuera y de fuera a dentro. El enfoque de dentro a fuera comienza a nivel de prueba de funciones individuales dentro de un programa.A menudo se denomina prueba de unidad, ya que las funciones podrían considerarse una unidad básica de computación, y llegaré a esto en la sección de soluciones. Empezaré con el enfoque de fuera a dentro.Esto significa que ejecutaré el programa desde la línea de comandos tal y como lo ejecutará el usuario. Se trata de un enfoque holístico para comprobar si las piezas del código pueden funcionar juntas para crear la salida correcta, por lo que a veces se denomina prueba de integración.
La primera de estas pruebas pasará la cadena de ADN como argumento de la línea de comandos y comprobará si el programa produce los recuentos correctos formateados en la cadena correcta:
def test_arg():
""" Uses command-line arg """
for file, expected in [TEST1, TEST2, TEST3]:  dna = open(file).read()
dna = open(file).read()  retval, out = getstatusoutput(f'{RUN} {dna}')
retval, out = getstatusoutput(f'{RUN} {dna}')  assert retval == 0
assert retval == 0  assert out == expected
assert out == expected 

Descomprime las tuplas en el
fileque contiene una cadena de ADN y el valorexpecteddel programa cuando se ejecuta con esta entrada.
Abre el archivo y lee el
dnadel contenido.
Ejecuta el programa con la cadena de ADN dada utilizando la función
subprocess.getstatusoutput()que me proporciona tanto el valor de retorno del programa como la salida de texto (también llamadaSTDOUT, que se pronuncia standard out).
Comprueba que el valor de retorno es
0, que indica éxito (o 0 errores).
Comprueba que la salida del programa es la cadena de números esperada.
La siguiente prueba es casi idéntica, pero esta vez pasaré el nombre del archivo como argumento al programa para comprobar que lee correctamente el ADN de un archivo:
def test_file():
""" Uses file arg """
for file, expected in [TEST1, TEST2, TEST3]:
retval, out = getstatusoutput(f'{RUN} {file}')  assert retval == 0
assert out == expected
assert retval == 0
assert out == expected

La única diferencia con respecto a la primera prueba es que paso el nombre del archivo en lugar de su contenido.
Ahora que ya has visto las pruebas, vuelve a ejecutarlas. Esta vez, utiliza pytest -xvdonde la bandera -v es para la salida verbosa. Dado que tanto -x como -v son banderas cortas, puedes combinarlas como -xv o -vx. Lee atentamente la salida y observa que intenta decirte que el programa está imprimiendo la secuencia de ADN, pero que la prueba espera una secuencia de números:
$ pytest -xv
============================= test session starts ==============================
...
tests/dna_test.py::test_exists PASSED [ 25%]
tests/dna_test.py::test_usage PASSED [ 50%]
tests/dna_test.py::test_arg FAILED [ 75%]
=================================== FAILURES ===================================
___________________________________ test_arg ___________________________________
def test_arg():
""" Uses command-line arg """
for file, expected in [TEST1, TEST2, TEST3]:
dna = open(file).read()
retval, out = getstatusoutput(f'{RUN} {dna}')
assert retval == 0
> assert out == expected  E AssertionError: assert 'ACCGGGTTTT' == '1 2 3 4'
E AssertionError: assert 'ACCGGGTTTT' == '1 2 3 4'  E - 1 2 3 4
E + ACCGGGTTTT
tests/dna_test.py:36: AssertionError
=========================== short test summary info ============================
FAILED tests/dna_test.py::test_arg - AssertionError: assert 'ACCGGGTTTT' == '...
!!!!!!!!!!!!!!!!!!!!!!!!!! stopping after 1 failures !!!!!!!!!!!!!!!!!!!!!!!!!!!
========================= 1 failed, 2 passed in 0.35s ==========================
E - 1 2 3 4
E + ACCGGGTTTT
tests/dna_test.py:36: AssertionError
=========================== short test summary info ============================
FAILED tests/dna_test.py::test_arg - AssertionError: assert 'ACCGGGTTTT' == '...
!!!!!!!!!!!!!!!!!!!!!!!!!! stopping after 1 failures !!!!!!!!!!!!!!!!!!!!!!!!!!!
========================= 1 failed, 2 passed in 0.35s ==========================

El
>al principio de esta línea indica que éste es el origen del error.
La salida del programa era la cadena
ACCGGGTTTTpero el valor esperado era1 2 3 4. Como no son iguales, se lanza una excepciónAssertionError.
Vamos a solucionarlo. Si crees que sabes cómo terminar el programa, por favor, salta directamente a tu solución. En primer lugar, prueba quizás a ejecutar tu programa para verificar que informa del número correcto de As:
$ ./dna.py A 1 0 0 0
Y luego Cs:
$ ./dna.py C 0 1 0 0
y así sucesivamente con Gsy Ts. Luego ejecuta pytest para ver si supera todas las pruebas.
Una vez que tengas una versión que funcione, considera la posibilidad de encontrar tantas formas diferentes como puedas de obtener la misma respuesta.Esto se llama refactorizar un programa. Tienes que empezar con algo que funcione correctamente, y luego intentar mejorarlo. Las mejoras pueden medirse de muchas formas. Quizás encuentres una forma de escribir la misma idea utilizando menos código, o quizás encuentres una solución que se ejecute más rápido. Independientemente de la métrica que utilices, sigue ejecutando pytest para asegurarte de que el programa es correcto.
Solución 1: Iterar y contar los caracteres de una cadena
Si no sabes por dónde empezar, trabajaré contigo en la primera solución. El objetivo es recorrer todas las bases de la cadena de ADN. Para ello, primero tengo que crear una variable llamada dna asignándole algún valor en el REPL:
>>> dna = 'ACGT'
Observa que cualquier valor encerrado entre comillas, ya sea simple o doble, es una cadena. Incluso un solo carácter en Python se considera una cadena. A menudo utilizo la función type() para verificar el tipo de una variable, y aquí veo que dna es de la clase str (cadena):
>>> type(dna) <class 'str'>
Escribe help(str) en el REPL para ver todas las cosas maravillosas que puedes hacer con cadenas.Este tipo de datos es especialmente importante en genómica, donde las cadenas constituyen gran parte de los datos.
En el lenguaje de Python, quiero recorrer los caracteres de una cadena, que en este caso son los nucleótidos del ADN.Un bucle for lo hará. Python ve una cadena como una secuencia ordenada de caracteres, y un bucle for visitará cada carácter de principio a fin:
>>> for base in dna:... print(base)
... A C G T

Cada carácter de la cadena
dnase copiará en la variablebase. Podrías llamar a estochar, ocpor carácter, o como quieras.
Cada llamada a
print()terminará con una nueva línea, por lo que verás cada base en una línea distinta.
Más adelante verás que los bucles for se pueden utilizar con listas y diccionarios y conjuntos y líneas de un archivo, básicamente cualquier estructura de datos iterable.
Contar los nucleótidos
Ahora que sé cómo visitar cada base de la secuencia, necesito contar cada base en lugar de imprimirla. Eso significa que necesitaré algunas variables para llevar la cuenta de los números de cada uno de los cuatro nucleótidos. Una forma de hacerlo es crear cuatro variables que contengan recuentos enteros, uno para cada base. Inicializaré cuatro variables para el recuento estableciendo sus valores iniciales en 0:
>>> count_a = 0 >>> count_c = 0 >>> count_g = 0 >>> count_t = 0
Podría escribir esto en una línea utilizando la sintaxis de desempaquetado de tuplas que mostré antes:
>>> count_a, count_c, count_g, count_t = 0, 0, 0, 0
Necesito mirar cada base y determinar qué variable debo incrementar, haciendo que su valor aumente en 1. Por ejemplo, si el base actual es una C, entonces debería incrementar la variable count_c. Podría escribir esto
for base in dna:
if base == 'C':  count_c = count_c + 1
count_c = count_c + 1 

El operador
==se utiliza para comparar dos valores para ver si son iguales. Aquí quiero saber si la cadena actualbasees igual a la cadenaC.
Establece
count_cigual a 1 mayor que el valor actual.
El operador == se utiliza para comparar dos valores para la igualdad. Funciona para comparar dos cadenas o dos números. Antes he mostrado que la división con / provocará una excepción si mezclas cadenas y números. ¿Qué ocurre si mezclas tipos con este operador, por ejemplo '3' == 3? ¿Es seguro utilizar este operador sin comparar antes los tipos?
Como se muestra en la Figura 1-3, una forma más breve de incrementar una variable utiliza el operador += para sumar lo que haya en el lado derecho (a menudo denominado RHS) de la expresión a lo que haya en el lado izquierdo (o LHS):
Figura 1-3. El operador += añadirá el valor del lado derecho a la variable del lado izquierdo
Como tengo cuatro nucleótidos que comprobar, necesito una forma de combinar tres expresiones más if. La sintaxis en Python para ello es utilizar elif para else if y else para cualquier caso final o por defecto. Aquí tienes un bloque de código que puedo introducir en el programa o en la REPL y que implementa un sencillo árbol de decisión:
dna = 'ACCGGGTTTT'
count_a, count_c, count_g, count_t = 0, 0, 0, 0
for base in dna:
if base == 'A':
count_a += 1
elif base == 'C':
count_c += 1
elif base == 'G':
count_g += 1
elif base == 'T':
count_t += 1
Debería acabar con recuentos de 1, 2, 3 y 4 para cada una de las bases ordenadas:
>>> count_a, count_c, count_g, count_t (1, 2, 3, 4)
Ahora tengo que informar del resultado al usuario:
>>> print(count_a, count_c, count_g, count_t) 1 2 3 4
Esa es la salida exacta que espera el programa.Observa que print() acepta múltiples valores para imprimir, e inserta un espacio entre cada valor. Si lees help(print) en la REPL, verás que puedes cambiar esto con el argumento sep:
>>> print(count_a, count_c, count_g, count_t, sep='::') 1::2::3::4
La función print() también pondrá una nueva línea al final de la salida, y esto también se puede cambiar utilizando la opción end:
>>> print(count_a, count_c, count_g, count_t, end='\n-30-\n') 1 2 3 4 -30-
Redactar y verificar una solución
Utilizando el código anterior, deberías ser capaz de crear un programa que supere todas las pruebas. Mientras escribes, te animo a que ejecutes regularmente pylint, flake8, y mypy para comprobar si tu código fuente contiene errores potenciales. Incluso iría más lejos y te sugeriría que instalaras las extensiones pytest para éstas, de modo que puedas incorporar rutinariamente dichas pruebas:
$ python3 -m pip install pytest-pylint pytest-flake8 pytest-mypy
Como alternativa, he colocado un archivo requirements.txt en el directorio raíz del repositorio de GitHub que enumera varias dependencias que utilizaré a lo largo del libro. Puedes instalar todos estos módulos con el siguiente comando:
$ python3 -m pip install -r requirements.txt
Con esas extensiones, puedes ejecutar el siguiente comando para ejecutar no sólo las pruebas definidas en el archivo tests/dna_test.py, sino también las pruebas de linting y comprobación de tipos mediante estas herramientas:
$ pytest -xv --pylint --flake8 --mypy tests/dna_test.py ========================== test session starts =========================== ... collected 7 items tests/dna_test.py::FLAKE8 SKIPPED [ 12%] tests/dna_test.py::mypy PASSED [ 25%] tests/dna_test.py::test_exists PASSED [ 37%] tests/dna_test.py::test_usage PASSED [ 50%] tests/dna_test.py::test_dies_no_args PASSED [ 62%] tests/dna_test.py::test_arg PASSED [ 75%] tests/dna_test.py::test_file PASSED [ 87%] ::mypy PASSED [100%] ================================== mypy ================================== Success: no issues found in 1 source file ====================== 7 passed, 1 skipped in 0.58s ======================
Algunas pruebas se omiten cuando una versión en caché indica que nada ha cambiado desde la última prueba. Ejecuta pytest con la opción ---cache-clear para forzar la ejecución de las pruebas. Además, puede que no superes las pruebas de linting si tu código no tiene el formato o la indentación adecuados. Puedes formatear automáticamente tu código utilizando yapf o black. La mayoría de IDEs y editores proporcionan una opción de autoformato.
Eso es escribir mucho, así que he creado un atajo para ti en forma de Makefile en el directorio:
$ cat Makefile
.PHONY: test
test:
python3 -m pytest -xv --flake8 --pylint --pylint-rcfile=../pylintrc \
--mypy dna.py tests/dna_test.py
all:
../bin/all_test.py dna.py
Puedes obtener más información sobre estos archivos leyendo el Apéndice A. Por ahora, basta con entender que si tienes make instalado en tu sistema, puedes utilizar el comando make test para ejecutar el comando en el objetivo test del Makefile. Si no tienes make instalado o no quieres utilizarlo, tampoco pasa nada, pero te sugiero que explores cómo se puede utilizar un Makefile para documentar y automatizar procesos.
Hay muchas formas de escribir una versión pasajera de dna.py, y me gustaría animarte a que sigas explorando antes de leer las soluciones. Más que nada, quiero que te acostumbres a la idea de cambiar tu programa y luego ejecutar las pruebas para ver si funciona. Éste es el ciclo del desarrollo dirigido por pruebas, en el que primero creo alguna métrica para decidir cuándo el programa funciona correctamente. En este caso, ése es el programa dna_test.py que ejecuta pytest.
Las pruebas garantizan que no me desvío del objetivo, y también me permiten saber cuándo he cumplido los requisitos del programa. Son las especificaciones (también llamadas specs) encarnadas en un programa que puedo ejecutar.¿De qué otra forma podría saber cuándo unprograma funciona o está terminado? O, como dice Louis Srygley: "Sin requisitos ni diseño, programar es el arte de añadir errores a un archivo de texto vacío".
Las pruebas son esenciales para crear programas reproducibles. A menos que puedas demostrar de forma absoluta y automática la corrección y previsibilidad de tu programa cuando se ejecuta con datos buenos y malos, entonces no estás escribiendo un buen software.
Soluciones adicionales
El programa que escribí anteriormente en este capítulo es la versión solution1_iter.py del repositorio de GitHub, así que no me molestaré en revisar esa versión. Me gustaría mostrarte varias soluciones alternativas que progresan de ideas más sencillas a otras más complejas. Por favor, no tomes esto como que progresan de peor a mejor. Todas las versiones superan las pruebas, así que todas son igualmente válidas. Se trata de explorar lo que Python puede ofrecer para resolver problemas comunes. Ten en cuenta que omitiré código que todas tienen en común, como la función get_args().
Solución 2: Crear una función count() y añadir una prueba unitaria
La primera variación que me gustaría mostrar trasladará todo el código de la función main() que realiza el recuento a una función count(). Puedes definir esta función en cualquier parte de tu programa, pero a mí generalmente me gusta get_args() en primer lugar, main() en segundo lugar, y luego otras funciones después de eso pero antes del pareado final que llama a main().
Para la siguiente función, también tendrás que importar el valor typing.Tuple:
def count(dna: str) -> Tuple[int, int, int, int]:""" Count bases in DNA """ count_a, count_c, count_g, count_t = 0, 0, 0, 0
for base in dna: if base == 'A': count_a += 1 elif base == 'C': count_c += 1 elif base == 'G': count_g += 1 elif base == 'T': count_t += 1 return (count_a, count_c, count_g, count_t)

Los tipos muestran que la función toma una cadena y devuelve una tupla que contiene cuatro valores enteros.

Este es el código de
main()que hizo el recuento.
Devuelve una tupla con los cuatro recuentos.
Hay muchas razones para trasladar este código a una función.Para empezar, se trata de una unidad de cálculo -dada una cadena de ADN, devuelve la frecuencia de tetranucleótidos-, así que tiene sentido encapsularlo. Esto hará que main() sea más corto y legible, y me permite escribir una prueba unitaria para la función. Como la función se llama count(), me gusta llamar a la prueba unitaria test_count().
He colocado esta función dentro del programa dna.py justo después de la función count() en lugar de en el programa dna_test.py por una cuestión de comodidad. Para programas cortos, tiendo a poner mis funciones y pruebas unitarias juntas en el código fuente, pero a medida que los proyectos se hacen más grandes, separaré las pruebas unitarias en un módulo aparte. Aquí tienes la función de prueba:
def test_count() -> None:""" Test count """ assert count('') == (0, 0, 0, 0)
assert count('123XYZ') == (0, 0, 0, 0) assert count('A') == (1, 0, 0, 0)
assert count('C') == (0, 1, 0, 0) assert count('G') == (0, 0, 1, 0) assert count('T') == (0, 0, 0, 1) assert count('ACCGGGTTTT') == (1, 2, 3, 4)

El nombre de la función debe empezar por
test_para quepytestpueda encontrarla. Los tipos que aparecen aquí muestran que la prueba no acepta argumentos y, como no tiene declaraciónreturn, devuelve el valor por defectoNone.
Me gusta probar las funciones con valores esperados e inesperados para asegurarme de que devuelven algo razonable. La cadena vacía debería devolver todos ceros.

El resto de las pruebas garantizan que cada base se informe en la posición correcta.
Para comprobar que mi función funciona, puedo utilizar pytest en el programa dna.py:
$ pytest -xv dna.py =========================== test session starts =========================== ... dna.py::test_count PASSED [100%] ============================ 1 passed in 0.01s ============================
La primera prueba pasa la cadena vacía y espera obtener todos ceros para los recuentos. Esto es una cuestión de juicio, sinceramente. Podrías decidir que tu programa debería quejarse al usuario de que no hay ninguna entrada. Es decir, es posible ejecutar el programa utilizando la cadena vacía como entrada, y esta versión informará de lo siguiente:
$ ./dna.py "" 0 0 0 0
Del mismo modo, si pasara un archivo vacío, obtendría la misma respuesta. Utiliza el comando touch para crear un archivo vacío:
$ touch empty $ ./dna.py empty 0 0 0 0
En los sistemas Unix, /dev/null es un filehandle especial que no devuelve nada:
$ ./dna.py /dev/null 0 0 0 0
Puedes considerar que ninguna entrada es un error e informar de ello como tal. Lo importante de la prueba es que me obliga a pensar en ello. Por ejemplo, ¿debería la función count() devolver ceros o lanzar una excepción si se le da una cadena vacía? ¿Debería el programa bloquearse con una entrada vacía y salir con un estado distinto de cero? Éstas son decisiones que tendrás que tomar para tus programas.
Ahora que tengo una prueba unitaria en el código de dna.py, puedo ejecutar pytest en ese archivo para ver si pasa:
$ pytest -v dna.py ============================ test session starts ============================= ... collected 1 item dna.py::test_count PASSED [100%] ============================= 1 passed in 0.01s ==============================
Cuando escribo código, me gusta escribir funciones que hagan sólo una cosa limitada con el menor número posible de parámetros.Entonces me gusta escribir una prueba con un nombre como test_ más el nombre de la función, normalmente justo después de la función en el código fuente.Si descubro que tengo muchas pruebas unitarias de este tipo, puede que decida moverlas a un archivo aparte y hacer que pytest ejecute ese archivo.
Para utilizar esta nueva función, modifica main() de la siguiente manera:
def main() -> None:
args = get_args()
count_a, count_c, count_g, count_t = count(args.dna)  print('{} {} {} {}'.format(count_a, count_c, count_g, count_t))
print('{} {} {} {}'.format(count_a, count_c, count_g, count_t)) 
Centrémonos por un momento en la función str.format() de Python. Como se muestra en la Figura 1-4, la cadena '{} {} {} {}' es una plantilla para la salida que quiero generar, y estoy llamando a la función str.format() directamente sobre un literal de cadena. Éste es un modismo común en Python que también verás con la función str.join().Es importante recordar que, en Python, incluso una cadena literal (una que existe literalmente dentro de tu código fuente entre comillas) es un objeto sobre el que puedes llamar a métodos.
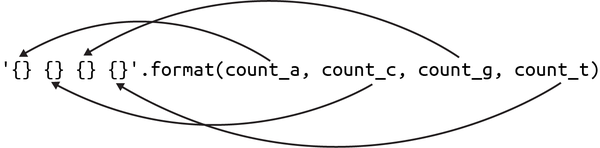
Figura 1-4. La función str.format() utiliza una plantilla que contiene llaves para definir marcadores de posición que se rellenan con los valores de los argumentos
Cada {} de la plantilla de cadena es un marcador de posición para algún valor que se proporciona como argumento a la función. Cuando utilices esta función, debes asegurarte de que tienes el mismo número de marcadores de posición que de argumentos. Los argumentos se insertan en el orden en que se proporcionan. Tendré mucho más que decir sobre la función str.format() más adelante.
No tengo que descomprimir la tupla devuelta por la función count(). Puedo pasar la tupla completa como argumento a la función str.format() si la descompongo añadiendo un asterisco (*) delante. Esto indica a Python que descomponga la tupla en sus valores:
def main() -> None:
args = get_args()
counts = count(args.dna)  print('{} {} {} {}'.format(*counts))
print('{} {} {} {}'.format(*counts)) 

La variable
countses una 4-tupla de los recuentos enteros de base.
La sintaxis
*countsexpandirá la tupla en los cuatro valores que necesita la cadena de formato; de lo contrario, la tupla se interpretaría como un único valor.
Como sólo utilizo la variable counts una vez, podría omitir la asignación y reducir esto a una línea:
def main() -> None:
args = get_args()
print('{} {} {} {}'.format(*count(args.dna))) 
Podría decirse que la primera solución es más fácil de leer y entender, y herramientas como flake8 podrían detectar cuándo el número de marcadores de posición {} no coincide con el número de variables. El código simple, verboso y obvio suele ser mejor que el código compacto e ingenioso. Aun así, es bueno saber sobre el desempaquetado de tuplas y el salpicado de variables, ya que utilizaré estas ideas en programas posteriores.
Solución 3: Utilizar str.count()
La función anterior count() resulta ser bastante verbosa. Puedo escribir la función con una sola línea de código utilizando el método str.count(). Esta función contará el número de veces que una cadena se encuentra dentro de otra cadena. Te lo mostraré en la REPL:
>>> seq = 'ACCGGGTTTT'
>>> seq.count('A')
1
>>> seq.count('C')
2
Si no se encuentra la cadena, informará 0, por lo que es seguro contar los cuatro nucleótidos aunque a la secuencia de entrada le falten una o más bases:
>>> 'AAA'.count('T')
0
Aquí tienes una nueva versión de la función count() utilizando esta idea:
def count(dna: str) -> Tuple[int, int, int, int]:""" Count bases in DNA """ return (dna.count('A'), dna.count('C'), dna.count('G'), dna.count('T'))
Este código es mucho más sucinto, y puedo utilizar la misma prueba unitaria para verificar que es correcto. Éste es un punto clave: las funciones deben actuar como cajas negras. Es decir, no sé ni me importa lo que ocurre dentro de la caja. Entra algo, sale una respuesta , y sólo me importa realmente que la respuesta sea correcta. Soy libre de cambiar lo que ocurre dentro de la caja siempre que el contrato con el exterior -los parámetros y el valor de retorno- siga siendo el mismo.
Aquí tienes otra forma de crear la cadena de salida en la función main() utilizando la sintaxis f-string de Python :
def main() -> None:
args = get_args()
count_a, count_c, count_g, count_t = count(args.dna)  print(f'{count_a} {count_c} {count_g} {count_t}')
print(f'{count_a} {count_c} {count_g} {count_t}') 

Descomprime la tupla en cada una de las cuatro cuentas.

Utiliza una cadena f para realizar una interpolación variable.
Se llama cadena f porque el f precede a las comillas. Utilizo el formato mnemotécnico para recordar que esto es dar formato a una cadena. Python también tiene una cadena sin formato que va precedida de un r, de la que hablaré más adelante. Todas las cadenas en Python -cadenas desnudas, f- o r- pueden ir entre comillas simples o dobles. No hay ninguna diferencia.
Con las cadenas f, los marcadores de posición {} pueden realizar interpolación de variables, que es una palabra de 50 centavos que significa convertir una variable en su contenido. Esos rizos pueden incluso ejecutar código. Por ejemplo, la función len() devolverá la longitud de una cadena y puede ejecutarse dentro de las llaves:
>>> seq = 'ACGT'
>>> f'The sequence "{seq}" has {len(seq)} bases.'
'The sequence "ACGT" has 4 bases.'
Normalmente, las cadenas f me parecen más fáciles de leer que el código equivalente que utiliza str.format(). Lo que elijas es sobre todo una decisión estilística. Yo recomendaría lo que haga que tu código sea más legible.
Solución 4: Utilizar un diccionario para contar todos los caracteres
Hasta ahora he hablado de las cadenas, las listas y las tuplas de Python. La siguiente solución introduce los diccionarios, que son almacenes de claves/valores.Me gustaría mostrar una versión de la función count() que utiliza internamente diccionarios para poder incidir en algunos puntos importantes que hay que entender:
def count(dna: str) -> Tuple[int, int, int, int]:""" Count bases in DNA """ counts = {}
for base in dna:
if base not in counts:
counts[base] = 0
counts[base] += 1
return (counts.get('A', 0),
counts.get('C', 0), counts.get('G', 0), counts.get('T', 0))

Internamente utilizaré un diccionario, pero no cambia nada de la firma de la función.

Inicializa un diccionario vacío para guardar el
counts.
Utiliza un bucle
forpara recorrer la secuencia.
Comprueba si la base aún no existe en el diccionario.

Inicializa el valor de esta base en
0.
Incrementa en 1 la cuenta de esta base.

Utiliza el método
dict.get()para obtener el recuento de cada base o el valor por defecto de0.
Una vez más, el contrato de esta función -la firma de tipo- no ha cambiado. Sigue siendo una cadena de entrada y una cuádruple de enteros de salida. Dentro de la función, voy a utilizar un diccionario que inicializaré utilizando las llaves vacías:
>>> counts = {}
También podría utilizar la función dict(). Ninguna de las dos es preferible:
>>> counts = dict()
Puedo utilizar la función type() para comprobar que se trata de un diccionario:
>>> type(counts) <class 'dict'>
La función isinstance() es otra forma de comprobar el tipo de una variable:
>>> isinstance(counts, dict) True
Mi objetivo es crear un diccionario que tenga cada base como clave y el número de veces que aparece como valor. Por ejemplo, dada la secuencia ACCGGGTTT, quiero que counts tenga este aspecto:
>>> counts
{'A': 1, 'C': 2, 'G': 3, 'T': 4}
Puedo acceder a cualquiera de los valores utilizando corchetes y un nombre de clave, así
>>> counts['G'] 3
Python lanzará una excepción KeyError si intento acceder a una clave de diccionario que no existe:
>>> counts['N'] Traceback (most recent call last): File "<stdin>", line 1, in <module> KeyError: 'N'
Puedo utilizar la palabra clave in para ver si una clave existe en un diccionario:
>>> 'N' in counts False >>> 'T' in counts True
Mientras itero por cada una de las bases de la secuencia, tengo que ver si existe una base en el diccionario counts. Si no existe, tengo que inicializarlo a 0. Entonces puedo utilizar con seguridad la asignación += para incrementar en 1 la cuenta de una base:
>>> seq = 'ACCGGGTTTT'
>>> counts = {}
>>> for base in seq:
... if not base in counts:
... counts[base] = 0
... counts[base] += 1
...
>>> counts
{'A': 1, 'C': 2, 'G': 3, 'T': 4}
Por último, quiero devolver una 4-tupla de los recuentos de cada una de las bases. Podrías pensar que esto funcionaría:
>>> counts['A'], counts['C'], counts['G'], counts['T'] (1, 2, 3, 4)
Pero pregúntate qué pasaría si faltara una de las bases de la secuencia. ¿Pasaría esto la prueba unitaria que escribí? Definitivamente no. Fallaría en la primera prueba utilizando una cadena vacía porque generaría una excepción KeyError. La forma segura de pedir un valor a un diccionario es utilizar el método dict.get().. Si la clave no existe, se devolverá None:
>>> counts.get('T')
4
>>> counts.get('N')
El método dict.get() acepta un segundo argumento opcional que es el valor por defecto a devolver cuando la clave no existe, por lo que ésta es la forma más segura de devolver una 4-tupla de las cuentas base:
>>> counts.get('A', 0), counts.get('C', 0), counts.get('G', 0),
counts.get('T', 0)
(1, 2, 3, 4)
Solución 5: Contar sólo las bases deseadas
La solución anterior contará todos los caracteres de la secuencia de entrada, pero ¿y si sólo quiero contar los cuatro nucleótidos?En esta solución, inicializaré un diccionario con valores de 0 para las bases deseadas. También necesitaré introducir typing.Dict para ejecutar este código:
def count(dna: str) -> Dict[str, int]:""" Count bases in DNA """ counts = {'A': 0, 'C': 0, 'G': 0, 'T': 0}
for base in dna:
if base in counts:
counts[base] += 1
return counts

La firma ahora indica que devolveré un diccionario que tiene cadenas para las claves y enteros para los valores.

Inicializa el diccionario
countscon las cuatro bases como claves y los valores de0.
Iterar por las bases.

Comprueba si la base se encuentra como clave en el diccionario
counts.
Si es así, incrementa en 1 la
countsde esta base.
Devuelve el diccionario
counts.
Como la función count() devuelve ahora un diccionario en lugar de una tupla, la función test_count() debe cambiar:
def test_count() -> None:
""" Test count """
assert count('') == {'A': 0, 'C': 0, 'G': 0, 'T': 0}  assert count('123XYZ') == {'A': 0, 'C': 0, 'G': 0, 'T': 0}
assert count('123XYZ') == {'A': 0, 'C': 0, 'G': 0, 'T': 0}  assert count('A') == {'A': 1, 'C': 0, 'G': 0, 'T': 0}
assert count('C') == {'A': 0, 'C': 1, 'G': 0, 'T': 0}
assert count('G') == {'A': 0, 'C': 0, 'G': 1, 'T': 0}
assert count('T') == {'A': 0, 'C': 0, 'G': 0, 'T': 1}
assert count('ACCGGGTTTT') == {'A': 1, 'C': 2, 'G': 3, 'T': 4}
assert count('A') == {'A': 1, 'C': 0, 'G': 0, 'T': 0}
assert count('C') == {'A': 0, 'C': 1, 'G': 0, 'T': 0}
assert count('G') == {'A': 0, 'C': 0, 'G': 1, 'T': 0}
assert count('T') == {'A': 0, 'C': 0, 'G': 0, 'T': 1}
assert count('ACCGGGTTTT') == {'A': 1, 'C': 2, 'G': 3, 'T': 4}

El diccionario devuelto siempre tendrá las claves
A,C,G, yT. Incluso para la cadena vacía, estas claves estarán presentes y se establecerán en0.
Todas las demás pruebas tienen las mismas entradas, pero ahora compruebo que la respuesta vuelve como un diccionario.
Cuando escribas estas pruebas, ten en cuenta que el orden de las claves en los diccionarios no es importante. Los dos diccionarios del código siguiente tienen el mismo contenido aunque se hayan definido de forma diferente:
>>> counts1 = {'A': 1, 'C': 2, 'G': 3, 'T': 4}
>>> counts2 = {'T': 4, 'G': 3, 'C': 2, 'A': 1}
>>> counts1 == counts2
True
Me gustaría señalar que la función test_count() prueba la función para asegurarse de que es correcta y también sirve como documentación. Leer estas pruebas me ayuda a ver la estructura de las posibles entradas y salidas esperadas de la función.
Así es como tengo que cambiar la función main() para utilizar el diccionario devuelto:
def main() -> None:
args = get_args()
counts = count(args.dna)  print('{} {} {} {}'.format(counts['A'], counts['C'], counts['G'],
print('{} {} {} {}'.format(counts['A'], counts['C'], counts['G'],  counts['T']))
counts['T']))
Solución 6: Utilizar collections.defaultdict()
Puedo librar a mi código de todos los esfuerzos anteriores para inicializar diccionarios y comprobar claves y demás utilizando la función defaultdict() del módulo collections:
>>> from collections import defaultdict
Cuando utilizo la función defaultdict() para crear un nuevo diccionario, le indico el tipo por defecto para los valores. Ya no tengo que comprobar una clave antes de utilizarla porque el tipo defaultdict creará automáticamente cualquier clave a la que haga referencia utilizando un valor representativo del tipo por defecto. Para el caso del recuento de nucleótidos, quiero utilizar el tipo int:
>>> counts = defaultdict(int)
El valor por defecto de int será 0. Cualquier referencia a una clave inexistente hará que se cree con un valor de 0:
>>> counts['A'] 0
Esto significa que puedo instanciar e incrementar cualquier base en un solo paso:
>>> counts['C'] += 1
>>> counts
defaultdict(<class 'int'>, {'A': 0, 'C': 1})
He aquí cómo podría reescribir la función count() utilizando esta idea:
def count(dna: str) -> Dict[str, int]:
""" Count bases in DNA """
counts: Dict[str, int] = defaultdict(int)  for base in dna:
counts[base] += 1
for base in dna:
counts[base] += 1  return counts
return counts

El
countsserá undefaultdictcon valores enteros.mypynecesita la anotación de tipo para asegurarse de que el valor devuelto es correcto.
Puedo incrementar con seguridad la
countspara esta base.
La función test_count() tiene un aspecto bastante diferente. Puedo ver a simple vista que las respuestas son muy distintas a las de las versiones anteriores:
def test_count() -> None:
""" Test count """
assert count('') == {}  assert count('123XYZ') == {'1': 1, '2': 1, '3': 1, 'X': 1, 'Y': 1, 'Z': 1}
assert count('123XYZ') == {'1': 1, '2': 1, '3': 1, 'X': 1, 'Y': 1, 'Z': 1}  assert count('A') == {'A': 1}
assert count('A') == {'A': 1}  assert count('C') == {'C': 1}
assert count('G') == {'G': 1}
assert count('T') == {'T': 1}
assert count('ACCGGGTTTT') == {'A': 1, 'C': 2, 'G': 3, 'T': 4}
assert count('C') == {'C': 1}
assert count('G') == {'G': 1}
assert count('T') == {'T': 1}
assert count('ACCGGGTTTT') == {'A': 1, 'C': 2, 'G': 3, 'T': 4}

Con una cadena vacía, se devuelve un diccionario vacío.

Observa que cada carácter de la cadena es una clave del diccionario.

Sólo está presente
A, con un recuento de 1.
Dado que el diccionario devuelto puede no contener todas las bases, el código de main() debe utilizar el método count.get() para recuperar la frecuencia de cada base:
def main() -> None:
args = get_args()
counts = count(args.dna)  print(counts.get('A', 0), counts.get('C', 0), counts.get('G', 0),
print(counts.get('A', 0), counts.get('C', 0), counts.get('G', 0),  counts.get('T', 0))
counts.get('T', 0))
Solución 7: Utilizar colecciones.Contador()
La perfección se alcanza, no cuando no hay nada más que añadir, sino cuando no queda nada que quitar.
Antoine de Saint-Exupéry
En realidad no me gustan mucho las tres últimas soluciones, pero necesitaba explicarte paso a paso cómo utilizar un diccionario tanto manualmente como con defaultdict() para que puedas apreciar la sencillez de utilizar collections.Counter():
>>> from collections import Counter
>>> Counter('ACCGGGTTT')
Counter({'G': 3, 'T': 3, 'C': 2, 'A': 1})
El mejor código es el que nunca escribes, y Counter() es una función preempaquetada que devolverá un diccionario con la frecuencia de los elementos contenidos en el iterable que le pases.También puedes oír que a esto se le llama bolsa o multiconjunto.Aquí el iterable es una cadena compuesta de caracteres, y así obtengo de vuelta el mismo diccionario que en las dos últimas soluciones, pero sin haber escrito código.
Es tan sencillo que prácticamente podrías prescindir de las funciones count() y test_count() e integrarlo directamente en tu main():
def main() -> None:
args = get_args()
counts = Counter(args.dna)  print(counts.get('A', 0), counts.get('C', 0), counts.get('G', 0),
print(counts.get('A', 0), counts.get('C', 0), counts.get('G', 0),  counts.get('T', 0))
counts.get('T', 0))

El
countsserá un diccionario que contendrá las frecuencias de los caracteres enargs.dna.
Sigue siendo más seguro utilizar
dict.get(), ya que no puedo estar seguro de que todas las bases estén presentes.
Podría argumentar que este código pertenece a una función count() y mantener las pruebas, pero la función Counter() ya está probada y tiene una interfaz bien definida. Creo que tiene más sentido utilizar esta función en línea.
Ir más lejos
Las soluciones aquí descritas sólo tratan secuencias de ADN proporcionadas como TEXTO EN MAYÚSCULAS. No es raro que estas secuencias se proporcionen en minúsculas. Por ejemplo, en genómica de plantas, es habitual utilizar bases en minúsculas para denotar regiones de ADN repetitivo. Modifica tu programa para que trate tanto la entrada en mayúsculas como en minúsculas haciendo lo siguiente:
-
Añade un nuevo archivo de entrada que mezcle los casos.
-
Añade una prueba a tests/dna_test.py que utilice este nuevo archivo y especifique los recuentos esperados insensibles a mayúsculas y minúsculas.
-
Ejecuta la nueva prueba y asegúrate de que tu programa falla.
-
Modifica el programa hasta que supere la nueva prueba y todas las pruebas anteriores.
Las soluciones que utilizaban diccionarios para contar todos los caracteres disponibles parecerían más flexibles. Es decir, algunas de las pruebas sólo tienen en cuenta las bases A, C, G y T, pero si la secuencia de entrada se codificara utilizando códigos IUPAC para representar la posible ambigüedad en la secuenciación, habría que reescribir el programa por completo. Un programa codificado para tener en cuenta sólo los cuatro nucleótidos tampoco serviría para las secuencias de proteínas que utilizan un alfabeto diferente. Considera la posibilidad de escribir una versión del programa que imprima dos columnas de salida con cada carácter que se encuentre en la primera columna y la frecuencia del carácter en la segunda. Permite al usuario ordenar ascendente o descendentemente por cualquiera de las dos columnas.
Revisa
Éste ha sido un capítulo un poco monstruoso. Los capítulos siguientes serán un poco más breves, ya que me basaré en muchas de las ideas fundamentales que he tratado aquí:
-
Puedes utilizar el programa
new.pypara crear la estructura básica de un programa Python que acepte y valide argumentos de la línea de comandos utilizandoargparse. -
El módulo
pytestejecutará todas las funciones cuyos nombres empiecen portest_e informará de los resultados de cuántas pruebas pasan. -
Las pruebas unitarias son para las funciones, y las pruebas de integración comprueban si un programa funciona como un todo.
-
Programas como
pylint,flake8ymypypueden encontrar varios tipos de errores en tu código. También puedes hacer quepytestejecute automáticamente pruebas para comprobar si tu código pasa estas comprobaciones. -
Los comandos complicados pueden almacenarse como objetivo en un Makefile y ejecutarse mediante el comando
make. -
Puedes crear un árbol de decisión utilizando una serie de declaraciones
if/else. -
Hay muchas formas de contar todos los caracteres de una cadena. Utilizar la función
collections.Counter()es quizá el método más sencillo para crear un diccionario de frecuencias de letras. -
Puedes anotar variables y funciones con tipos, y utilizar
mypypara asegurarte de que los tipos se utilizan correctamente. -
El REPL de Python es una herramienta interactiva para ejecutar ejemplos de código y leer documentación.
-
La comunidad Python suele seguir directrices de estilo como PEP8. Herramientas como
yapfyblackpueden formatear automáticamente el código según estas sugerencias, y herramientas comopylintyflake8informarán de las desviaciones de las directrices. -
Las cadenas, listas, tuplas y diccionarios de Python son estructuras de datos muy potentes, cada una con métodos útiles y abundante documentación.
-
Puedes crear un tipo personalizado e inmutable
classderivado de tuplas con nombre.
Tal vez te preguntes cuál es la mejor de las siete soluciones. Como ocurre con muchas cosas en la vida, depende. Algunos programas son más breves de escribir y más fáciles de entender, pero pueden no funcionar bien cuando se enfrentan a grandes conjuntos de datos. En el Capítulo 2, te mostraré cómo comparar programas, enfrentándolos entre sí en múltiples ejecuciones utilizando grandes entradas para determinar cuál funciona mejor.
1 Los tipos booleanos son True o False, pero muchos otros tipos de datos son verdaderos o, a la inversa, falsos. La cadena vacía str ("") es falsa, por lo que cualquier cadena no vacía es verdadera. El número 0 es falso, por lo que cualquier valor distinto de cero es verdadero. Un list, set o dict vacío es falso, por lo que cualquiera de ellos que no sea vacío es verdadero.
Get Dominar Python para Bioinformática now with the O’Reilly learning platform.
O’Reilly members experience books, live events, courses curated by job role, and more from O’Reilly and nearly 200 top publishers.

